生物信息学笔记
第三章 序列比较
3.1 认识序列
序列分类
蛋白质序列和核酸序列。蛋白质序列是由 20 个不同的字母,也就是 20 种不同的氨基酸排列组合而成。核酸序列是由 4 种不同的字母,也就是 4 种 不同的碱基排列组合而成。核酸序列又分为 DNA 序列和 RNA 序列。
序列格式
生物序列有自己的书写格式,而且格式很多。不同的处理软件会用到不同的格式,但是最常用的,大多数软件都识别的格式是 FASTA 格式。
FASTA 格式,第一行是一个大于号“>”开头,后面紧接注释信息,比如序 列的名字,编号等。因为前面有大于号,序列分析软件会自动识别这是一条序列的开始,且 这一行是注释而不是序列。从第二行开始就是纯序列部分。
3.2 序列相似性
序列相似性的意义
意义在于,相似的序列往往起源于一个共同的祖先。它们很可能有相似的 空间结构和生物学功能。因此,对于一个已知序列但未知结构和功能的蛋白质,如果与它序 列相似的某些蛋白质的结构和功能已知,那么就可以推测出这个未知结构和功能的蛋白质的结构和功能。简言之,相似的序列意味着相似的结构,相似的结构意味着相似的功能
一致度和相似度
一致度可以定义为它们对应位置上相同的残基数目占总长度的百分比。一个残基就是指一个字母(氨基酸或碱基)
相似度可以暂时定义为他们对应位置上相似的残基 与相同的残基的数目和占总长度的百分比
3.3 替换计分矩阵
核酸序列的替换记分矩阵
等价矩阵
这个矩阵最简单. 其中,相同核苷酸之间的匹配得分为 1,不同核苷酸间的替换得分为 0。由于不含有碱基的 理化信息和不区别对待不同的替换,在实际的序列比较中很少使用,一般只用于理论计算。
转换-颠换矩阵
我们说 核酸的碱基按照环结构特征被划分为两类,一类是嘌呤,包括腺嘌呤 A 和鸟嘌呤 G,它们都有两个环;另一类是嘧啶,包括胞嘧啶 C 和胸腺嘧啶 T,它们只有一个环。
如果 DNA 碱基的替换保持环数不变,则称为转换,比如腺嘌呤 A 替换为 鸟嘌呤 G、或者胞嘧啶 C 替换 为胸腺嘧啶 T,也就是嘌呤变嘌呤,嘧啶变嘧啶;如果环数发生变化,则称为颠换,比如腺 嘌呤 A 替换为胞嘧啶 C、或者胸腺嘧啶 T 替换为鸟嘌呤 G,也就是嘌呤变嘧啶,或者嘧啶 变嘌呤。
在进化过程中,转换发生的频率远比颠换高。也就是说,大自然更倾向于接受嘌呤 和嘌呤之间的替换,以及嘧啶和嘧啶之间的替换,而嘌呤和嘧啶之间的替换会导致不好的事 情发生,这种替换大多在进化过程中已经被淘汰了。为了反映这一情况,转换-颠换矩阵中, 转换的得分比颠换要高为-1 分,而颠换的得分为-5 分。
BLAST矩阵
经过大量实际比对发现,如果令被比对的两个核苷酸相同时 得分为+5 分,不相同为-4 分,这时比对效果最好。这个矩阵广泛地被 DNA 序列比较所采 用。没有为什么,就是好,实践经验所得。因为这个矩阵最早应用于 BLAST 工具,因此得名 BLAST 矩阵

蛋白质序列的替换记分矩阵
等价矩阵
PAM 矩阵
PAM 矩阵基于进化原 理。如果两种氨基酸替换频繁,说明自然界容易接受这种替换,那么这一对氨基酸替换的得 分就应该高。
PAM 矩阵是目前蛋白质序列比较中最广泛使用的记分方法之一。基础的 PAM-1 矩阵反应的是进化产生的每一百个氨基酸平均发生一个突变的量值,由统计方法得到。 PAM-1 自乘 n 次,可以得到 PAM-n ,表示发生了更多次突变。
我们需要根据要比较的序列之间的亲缘关系远近,来选择适合的 PAM 矩阵。如果序列亲缘关系远,也就是说序列间会 有很多突变,那就选 PAM 后面跟一个大数字的矩阵。如果亲缘关系近,也就是突变比较少, 序列间大多数地方都是一样的,那就选 PAM 后面跟一个小数字的矩阵。
如PAM250矩阵。对角线上的数值为匹配氨基酸的得分。其他位置上≥0 的得分代 表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。

BLOSUM 矩阵
BLOSUM 矩阵有和 PAM 矩阵相同的地方,也有不同的地方。相同的是,BLOSUM 矩阵 后面也带有一个编号,有很多种 BLOSUM 矩阵。不同的是,BLOSUM 矩阵都是通过对大量 符合特定要求的序列计算而来的。这点和 PAM 矩阵不同的。
PAM-1 矩阵是基于相似度大于85%的序列计算产生的,也就是通过关系较近的序列计算出来的。那些进化距离较远的矩阵, 如 PAM-250,是通过 PAM-1 自乘得到的。也就是说,BLOSUM 矩阵的相似性是根据真实数 据产生的,而 PAM 矩阵是通过矩阵自乘外推而来的。和 PAM 矩阵的另一个不同之处是 BLOSUM 矩阵的编号。这些编号,比如 BLOSUM80 中的 80,代表这个矩阵是由一致度≥80% 的序列计算而来的。同理,BLOSUM62 是指这个矩阵是由一致度≥62%的序列计算而来的。 因此,BLOSUM 后面跟一个小数字的矩阵适合用于比较相似度低的序列,也就是亲缘关系 远的序列;而 BLOSUM 后面跟一个大数字的矩阵适合比较相似度高的序列,也就是亲缘关 系近的序列。
BLOSUM 62 矩阵.样子和 PAM 矩阵差不多,但是里面的数值是不一样的。同样, ≥0 的得分代表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。

亲缘关系较近的序列之间的比较,用 PAM 数小的矩阵或 BLOSUM 数大的矩阵;而亲缘关系较远的序列之间的比较,用 PAM 数大的矩阵或 BLOSUM数小的矩阵。

遗传密码矩阵
它是通过计算一个氨基酸转换成另一个氨基酸所需的密码子变化的数目而得到的。 矩阵的值对应为据此付出的代价。如果变化一个碱基就可以使一个氨基酸的密码子转换为另 一个氨基酸的密码子,则这两个氨基酸的替换代价为 1;如果需要 2 个碱基的改变,则替换 代价为 2;再比如从蛋氨酸(Met)到酪氨酸(Tyr)三个密码子都要变,则代价为 3。
遗传密码矩阵常用于进化距离的计算,它的优点是计算结果可以直接用于绘制进化树,但是它在 蛋白质序列比对,尤其是相似程度很低的蛋白质序列比对中,很少被使用。

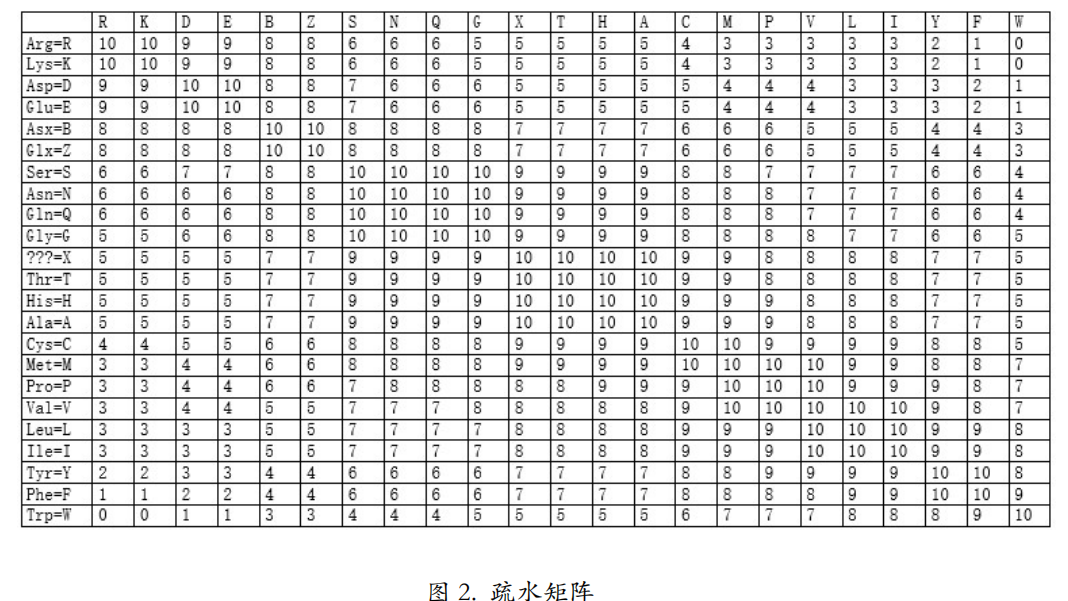
疏水矩阵
它是根据氨基酸残基替换前后疏水性的变化而得到的矩阵。 若一次氨基酸替换导致疏水特性不发生太大的变化,则这种替换得分高,否则替换得分低。 疏水矩阵物理意义明确,有一定的理化性质依据,适用于偏重蛋白质功能方面的序列比对。 在这个矩阵里,氨基酸按照亲疏水性排列。前边是亲水的,后面是疏水的。
3.4 序列比对-Dotplot打点法
序列比对概念
Sequence aligment:运用特定的算法找出两个或者多个序列之间产生最大相似度得分的空格插入和序列排列方案。
比如:
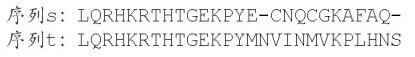
这两条序列的比对就是把 s 和 t 这两个字符串上下排列起来,在某些位置插入空格,这些空格叫空位(gap)。然后依次比较它们在每一个位置上字符的匹配情况,匹配的好,这个位置 就会得高分,匹配的不好,看看能不能左右错一错,或填上个空位,让附近的位置更好的匹 配在一起,从而使所有位置的得分之和尽可能的高。说白了,就是通过插入空位,让上下两行中尽可能多的一致的和相似的字符对在一起。这不是随便摆摆看看就能完成的,需要使用专门的序列比对算法。
根据比对序列的个数可以把序列比对分为双序列比对和多序列比对。顾名思义,双序列 比对就是比 2 条,而多序列比对是比 2 条以上。此外根据序列比对的算法不同,
双序列比对 又分为全局比对和局部比对。全局比对就是全长比较,一个字符都不能落下。而局部比对是 比较对得最好的局部,对得不好的部分会被忽略不计。我们将从双序列比对的全局比对入手,看看序列比对的算法是如何插入空位,并使得插入后两条序列的得分最高的。
打点法概念
打点法是最简单的比较两个序列的方法,理论上可以用纸和笔来完成。如果要比较下面这两条序列:

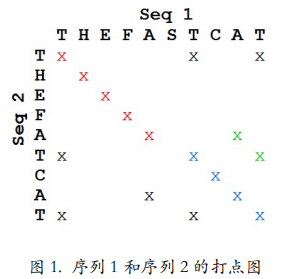
在打点矩阵中,连续的对角线及对角线的平行线代表两条序列中相同的区域。
这个矩阵 中在主对角线位置上连续的红色的对角线说明这个位置对应的序列 1 的部分和序列 2 的部分是完全相同的,都是 THEFA。此外,跟红对角线平行的蓝色平行线和绿色平行线,同样指出了序列 1 和序列 2 中两条相同的序列。也就是序列 1 和序列 2 中对应位置的 TCAT,以及序列 1 和序列 2 中对应位置的 AT。由这三条线,我们找到了序列 1 和序列 2 中三条相同的 子序列。最后,我们放眼全局,红色的线和蓝色的线加起来基本上构成了一条主对角线。由 此我们可以得出结论:序列 1 和序列 2 是比较相似的两条序列
除了可以用打点法给两条不同的序列打点,还可以用一条序列自己跟自己打点。这样可 以发现序列中重复的片段。比如我们让下面这条序列自己和自己打点:


这样的打点矩阵必然是对称的,并且一定有一条主对角线(图 3)。此外,在横向或纵向上, 与主对角线平行的短平行线所对应的序列片段就是重复的部分。其中,红色短平行线对应的 ‘THE’在序列中重复出现了 3 次。包括主对角线在内,平行线出现的次数就是重复的次数。
用这种方法我们还可以快捷的发现序列中的串联重复序列以及重复的次数。我们只要数数在半个矩阵中包括主对角线在内的所有等距的平行线的个数,就可以知道重复的次数,而且最短的平行线对应的序列就是重复单元。
短的串联重复序列具有高度多态性,也就 是说不同的个体间重复次数存在差异,而且这种差异在基因遗传过程中一般遵循孟德尔共显性遗传规律,所以快速查找某些特定的短的串联复序列的重复次数可以用于法医学的个体识别或亲子鉴定等领域。
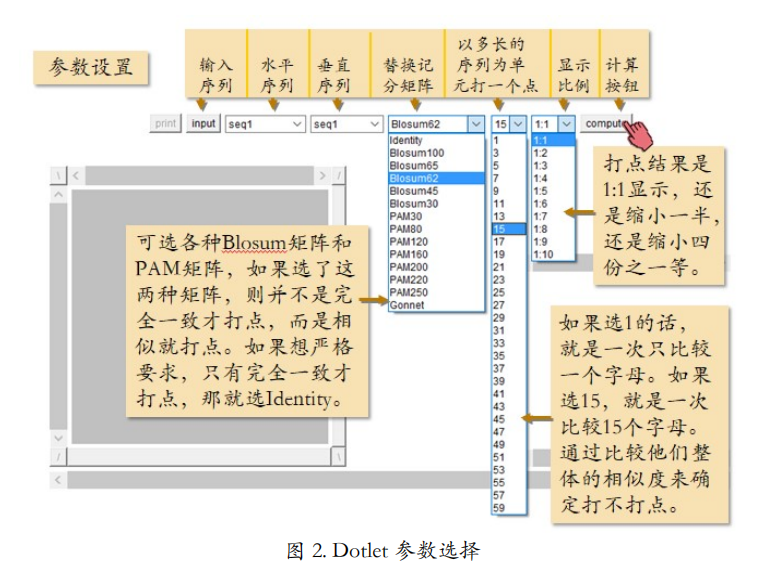
Dotlet 界面介绍
可以自动打点的软件有很多(表 1)。我们挑其中最常用的 Dotlet 软件做为演示(http://myhits.isb-sib.ch/cgi-bin/dotlet。Dotlet 基于 Java 开发,所以页面打开后会蹦出 JAVA 对话框。像对待 Jsmol 一样,接受 JAVA,信任 JAVA,运行 JAVA。当然前提是你的电脑已 经安装了 JAVA。如果还没有安装,可以到课程附件或者 JAVA 官网下载安装。别忘了安装后,重启浏览器,JAVA 才能生效。同样的,IE 如果不好使,可以尝试其他浏览器。

| 名称 | 网址链接 |
|---|---|
| Dotlet | http://myhits.isb-sib.ch/cgi-bin/dotlet |
| Dnadot | http://arbl.cvmbs.colostate.edu/molkit/dnadot |
| Dotter | http://sonnhammer.sbc.su.se/Dotter.html |
| Dottup | http://emboss.sourceforge.net |
Dotlet 应用实例
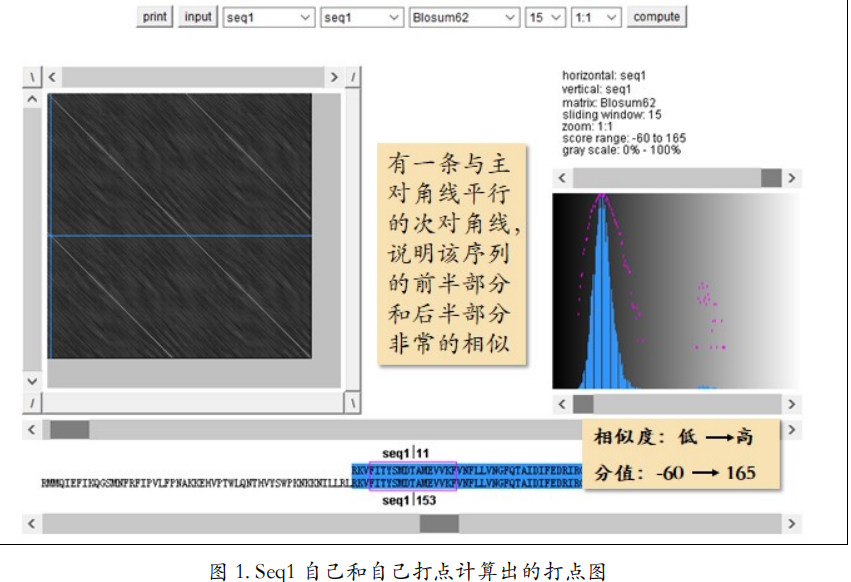
图 1 是上一节中 seq1 自己和自己打点计算出的打点图。注意默认的颜色方案是在越相 似的地方打的点的颜色越浅,越不相似的地方颜色越深。所以整体感觉像是在一张黑纸上打 白点。因为是 seq1 自己和自己打点,所以应该有一条明显的主对角线,这是所有自己和自 己打点的序列都会出现的情况。除此之外,还有一条与主对角线平行的次对角线,涉及 seq1 大约 1/2 的长度。这说明 seq1 的前半部分和后半部分非常的相似!
我们把鼠标点在这条次对角线上的任意位置,之后就可以从下面的序列显示区域看到,这一点,对应横着这条 seq1 的位置 153 开始的这一段,同时对应竖着这条 seq1 的位置 11 开 始的这一段。为什么这一点对应的是一段呢?因为我们这里选的单元长度是 15,也就是一 次比较 15 个字母。从这里也可以看出,如果把 seq1 的前半段和后半段重合起来的话,它们是完全一样的序列。
图 1 右边给出了打点所用的参数设置。按照选定的替换计分矩阵和单元长度计算,比较 所能打出的最高分是 165 分,最低分是-60 分。默认的颜色方案是灰度从 0%到 100%。也就 是最高分 165 分的点具有 100%的灰度,即纯白色;最低分-60 分具有 0%的灰度,即纯黑色。参数下面的图给出了各个分值的点的个数。横坐标是分值,从-60 到 165,纵坐标是得某一分值的点的个数。我们可以看到绝大多数点的分值都是较低的,也就是绝大多数位置的比较 结果都是不相似的。只有少数对角线上的比较才是高分值的点,因为这些位置对应的是相似 的区域。这个情况反映在打点图上就是,绝大多数点都是深色的,只有少数点是浅色的,这 些浅色的点位于主对角线和两条对称的次对角线上。
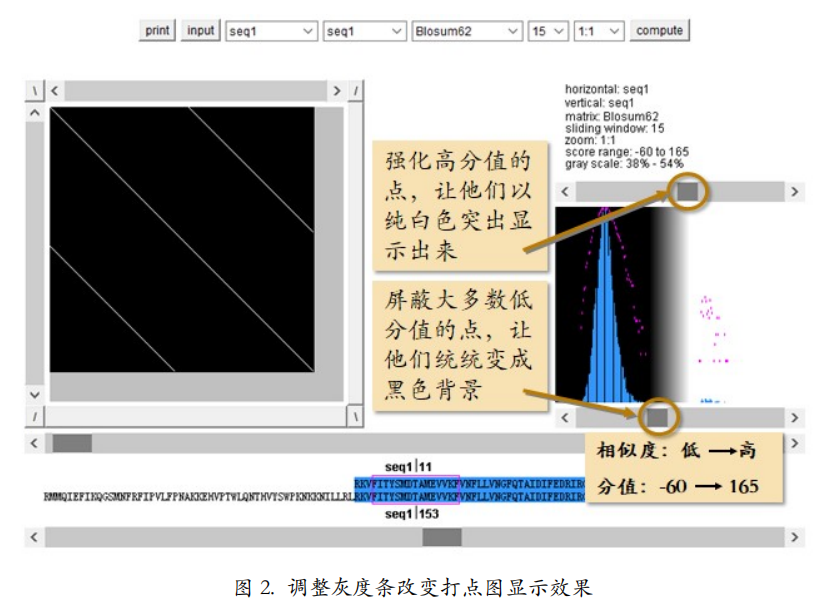
这里我们可以通过调整灰度条,来屏蔽大多数低分值的点,让他们统统变成黑色背景, 并且强化高分值的点,让他们以纯白色突出显示出来(图 2)。
3.5 序列比对-动态规划法
双序列全局比对
经典的全局比对算法是 Needleman-Wunsch 算法。1970 年,Needleman 和 Wunsch 首先将动态规划法应用于两条序列的全局比对,后来这个算法就称为 Needleman-Wunsch 算法。今天,所有比对软件使用的算法都是从这个经典算法衍生出来的。
我们用 Needleman-Wunsch 算法为序列 p 和序列 q 创建全局比对。输入值除了两条序列 之外,还要有替换计分矩阵以确定不同字母间的相似度得分,以及空位罚分(图 1)。空位 罚分就是当字母对空位的时候应该得几分。我们还是希望一致或相似的字母尽可能的对在一 起,字母对空位的情况和不相似的字母对在一起的情况一样,都不是我们希望的,还是少出 现为好,所以通常字母对空位会得到一个负分,这个负分就叫做空位罚分。这里我们让空位罚分,也就是 gap 分值为-5 分。在比对中没有空位对空位的情况。输入值就是这些。
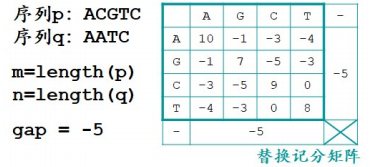
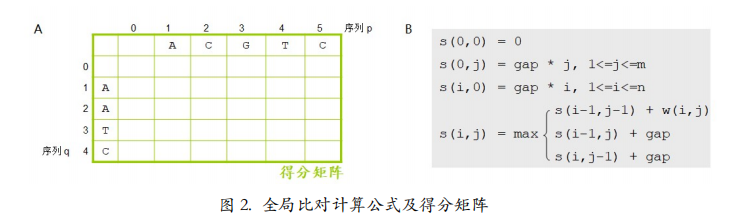
接下来我们要创建一个得分矩阵(图 2-1),并根据公式(图 2-2)把得分矩阵填满。填 满后全局比对就会跃然于纸上。得分矩阵的第一行是序列 p,第一列是序列 q,这一步和打点法很像。不过要注意,p 和 q 的前面各留一个空列和一个空行,也就是第 0 列和第 0 行。
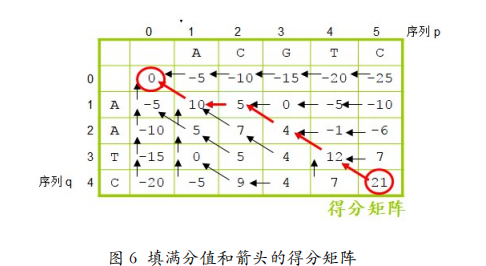
按照上面的公式,将整个得分矩阵填满。这时,我们再回过头来看一下第一行和第一列 (图 6)。其实,第一行的每一个值都是从左边的格加 gap 来的。所以我们给它们补上向左 的箭头。第一列的每一个值都是从上边的格加 gap 来的。所以我们给它们补上向上的箭头。 至此,所有的箭头和数值就都填好了。填满之后,右下角的分数就是整个全局比对最终的得 分。然后从这个位置开始追溯箭头一直到左上角的零,并且把这些箭头标记出来。
图 6 中标出的红色箭头是写出全局比对的唯一依据。追溯箭头是从右下角到左上角,但是写全局比对是从左上角开始,如果是斜箭头则是字符对字符,如果是水平箭头或垂直箭头 则是字符对空位,箭头指着的序列为空位。我们看第一个是斜箭头,字母对字母,就是 A 对 A,第二个是水平箭头,字母对空位,箭头指着的序列是空位,也就是 C 对空位。然后 斜箭头 G 对 A,斜箭头 T 对 T,斜箭头 C 对 C,一直写到右下角,全局比对就出现了(图 7)。 唯一的一个空位插在序列 q 的 A 与 A 之间,这样最终的比对得分最高。不信的话可以试试, 其他任何一种插入空位的比对结果,得分都不会超过 21 分。因为我们在得分矩阵的创建过 程中,每一步都是在上一步最优的情况下得出的当前最优结果。

双序列局部对比
在看局部比对的算法之前,先看看序列 a 和序列 b 这两条序列,谁和序列 c 更相似一些 (图 1)。AC 做全局比对的得分是-46 分。BC 做全局比对的得分是 8 分。单从分数上看,b 跟 c 更相似,因为得分高。但是如果只看红框里这部分,a 和 c 显然比 b 和 c 对得要好。如 果只比较红框里这部分的话,a 和 c 的得分是 24 分,比 b 和 c 的得分要高。这个例子告诉我 们,对于像这样一长一短的两条序列,比较局部比比较全长更有意义。这就是为什么除了全局比对,还有局部比对。

局部比对的算法和全局比对很相似,只是在选最大值时通过增加了第四个元素“0”,来 达到比对局部的效果。序列 p 和序列 q,一长一短,其他输入值跟全局比对的一样(图 2)。
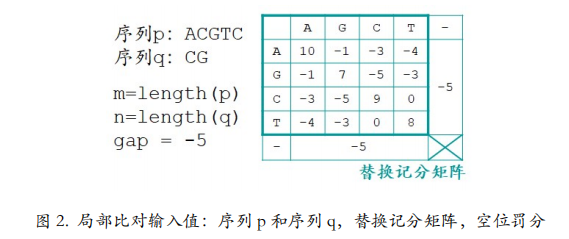
局部比对的计算公式在全局比对的基础上增加了第四个元素“0”。得分矩阵初始值仍是 0,但第一行和第一列与全局比对不同,全是 0(图 3)。
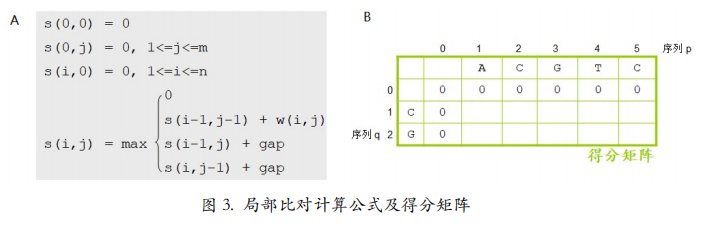
从 s(1,1)开始要选择四个值中的最大值。除了上面格 s(0,1)+gap=0+-5=-5,左边 格 s(1,0)+gap=0+-5=-5,斜上格 s(0,0)+w(1,1)=0+-3=-3,还有一个 0。max(-5, -5,-3,0)=0。并且这个 0 既不是从上面格,也不是从左边格,以及斜上格三个方向来的, 而是来自于公式里增加的“0”,所以不用画箭头(图 4)。
s(1,4)的计算:上面格 s(0,4)+gap=0+-5=-5,左边格 s(1,3)+gap=4+-5=-1, 斜上格 s(0,3)+w(1,4)=0+0=0,还有一个 0。max(-5,-1,0,0)=0。这个 0 和 s(1,1) 的 0 是不一样的。这个 0 应该画上斜上的箭头(图 5),因为它可以来自公式中的 0,也可 以来自斜上格。而 s(1,1)的 0 没有箭头因为它只来自公式中的 0。两种情况虽然都是 0, 但来源不同,一定要通过箭头标识清楚。
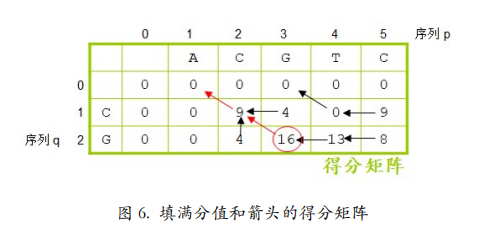
按照公式,填充满整个得分矩阵(图 6)。与全局比对不同,局部比对的得分不是在右下角,而是在整个矩阵中找最大值。这个最大值才是局部比对的最终得分,他可能出现在任何一个位置。这次箭头追溯也不是从右下角到左上角,而是从刚刚找到的最大值开始追溯到没有箭头为止。追溯箭头终止的位置也可以是得分矩阵中的任何一个位置。
最后根据标记好的箭头写出比对结果(图 7)。从左上到右下标记的红色箭头依次是: 斜箭头字母对字母,C 对 C;斜箭头字母对字母,G 对 G。相比这两条序列的全局比对结果, 两端的空位在局部比对中就全部被忽略掉了。

3.6 一致度和相似度
我们回过头来,再看看一致度和相似度的计算。之前我们卡在长度不一样的两个序列如 何计算一致度和相似度这个问题上。现在学会了序列比对,我们就可以先给这两条长度不同 的序列做全局比对,然后计算全局比对中一致字符的个数和相似字符的个数,再除以全局比对的长度,就可以得到它们的一致度和相似度了。比如下面这两条序列:
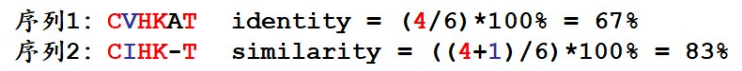
首先做出它们的全局比对,比对中一致字符的个数是 4 个,全局比对长度 6,一致度=67%。 相似字符个数 1,相似度就是(4+1)/6=83%。
由此,我们必须回过头来,把长度相同的两个序列计算一致度和相似度的方法重新规范 一下。尽管长度相同,但是做出的全局比对的长度并不一定等于序列的长度,比如下面这两条序列:
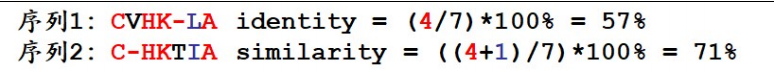
上下各加入一个空位,全局比对的长度就不等于序列的长度了。所以不管两条序列长度是否相同,都要先对它们做全局比对。让两条序列先以最优的方式比对起来,再从全局比对中数 出一致字符和相似字符的个数,除以全局比对的长度,来得到它们的一致度和相似度。
3.7 在线双序列比对工具
EMBL全局双序列比对工具
目前,使用率最高的是 EMBL 网 站的双序列比对工具(http://www.ebi.ac.uk/Tools/psa)。打开页面,上面有全局比对工具、 局部比对工具、还有基因组比对工具。
首先看全局比对中的蛋白质序列比对工具(图 1)。输入值非常简单,把要比较的两条 蛋白质序列贴在输入框里或者上传。可以使用示例文件 global.fasta 里面的两条序列。如果想 要进一步设置比对的参数,可以点 More options。从这里可以选择使用哪种替换记分矩阵。 按照之前讲过的原则,选择 PAM 矩阵或 BLOSUM 矩阵。如果实在不知道选哪个矩阵,就 闭着眼睛选 BLOSUME62 吧!下拉菜单里默认选的就是BLOSUM62。除了选择替换记分矩 阵,这里还可以设置空位罚分,也就是 gap 的分值。这里实际上是让你选空位对字母的情况罚几分,所以显示的是正数,但在计算的过程中还是按照负数处理。
计算机只需要用几秒钟的时间,就可以计算出结果(图 2)。在结果页面里,上部是比对使用的参数,以及得出的序列比对的长度、一致度、相似度、空位比例和比对得分。下部就是序列比对。在序列比对里,左边是两条序列的名字,因为输入的是 FASTA 格式的序列, 所以程序自动识别出了序列的名字。右边的序列比对分 3 行。上下两行是序列,里面插入了 许多的空位。中间这行标记出了哪些位置上下两个字母是完全相同的,用竖线表示。上下两个字母相似,用双点表示。上下不相似,用单点表示。字母对空位的情况,用空格表示。
这样,我们只要数数比对结果里竖线的个数(40 个),再除以比对的长度(196 个),就可计算 出一致度。再用竖线的个数加上双点的个数(40+29=69 个),除以比对长度(196 个),就是相似度。整个比对里一共插入了 65 个空位,占整个比对长度的 33%。序列两边的数是这一行中的字母在序列中的位置数,而不是这一行的长度。比如第二行是 seq1 的第 49 个字母 到第 97 个字母,是 seq2 的第 27 个字母到第 75 个字母。

EMBL 局部双序列比对工具
EMBL 的局部双序列比对工具可以选择经典的 Smith-Waterman 算法。仍然比对蛋白质序 列。输入的序列在示例文件 local.fasta 里面。More options 里面的参数设置和全局比对是一样 的。在这个例子里,我们保持所有参数都为默认值,点提交。
从比对结果可以看出(图 2),只有中间黑色的相似的部分出现在比对结果中了,两头 红色的不相似的部分被忽略掉了。也就是只返回了局部最相似,得分最高的片段的比对结果。

用这两条序列再做一次全局比对(图 3)。从两次的比对结果可以更清楚的看出,全局比对里前面和后面对得不好的部分在局部比对里就都被忽略了

除了一长一短两条序列适合做局部比对,有的时候两条差不多长的序列也可以做局部比对,以找出它们最相似的局部片段。比如示例文件 local2.fasta 里面的两条序列长度差不多。我们做局部比对看看结果如(图 4)。为了让相似的部分突出出来,我们把 gap 都调大,gap开头调到 10,gap 延长调到 5,提交。
比对结果中,只有黑色的相似的部分出现在最终的比对结果中了,两头红色的不相似的 、部分全部被忽略了(图 5)。

如果给这两条序列做全局比对的话,会发现,绝大部分位置对得都很差,只有中间这一 段对的还不错(图 6)。所以,有时候两条序列并不同源,它们只是有一个功能相似的区域,这时用局部比对我们就能很快找到这一区域在两条序列中的位置。但是如果做全局比对的话,结果就不如局部比对明显了。

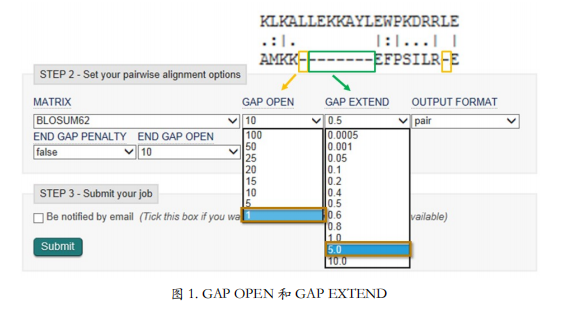
Gap 的类型及分值设置
EMBL 比对工具将 gap 分为两种,一种叫“gap 开头(GAP OPEN)”,另一种叫“gap 延长(GAP EXTEND)”(图 1)。gap 开头就是连续的一串 gap 里面打头的那一个,可以当 它是队长。gap 延长就是剩下的那些 gap,也就是队长后面跟着的小兵。这一串里,第一个 gap 是 gap 开头,后面的都是 gap 延长。单独的一个 gap 按 gap 开头算。
gap 开头和 gap 延长 可以分别定义不同的罚分。默认情况下,gap 开头罚分多,gap 延长罚分少。全局比对的例子里我们就是用这种搭配组合方案做出的比对。这次我们反过来试试,让 gap 开头罚分少, 让 gap 延长罚分多。比如 gap 开头选罚 1 分,gap 延长选罚 5 分,其他参数不变,再作一次看看结果发生了什么变化。
当 gap 开头小,gap 延长大的时候,做出来的比对里面,gap 很分散,极少有连续长串 的 gap 出现(图 2-A)。开头的一串 gap 是个例外,因为 seq2 太短, seq1 的这一段只能跟 gap 相对。其他部分的 gap 都是分散出现的。这和我们第一次做出来的比对结果是截然不同的(图 2-B)。在第一次做的结果里,也就是 gap 开头大,gap 延长小的时候,gap 很集中,有很多成 长串出现的 gap。
大家可以想想其中的奥妙。当 gap 开头大,gap 延长小的时候,说明在连续的字母里插入一个 gap 打开一个缺口要付出很大的代价,因为 gap 开头罚分大。但是这个 缺口一旦打开了,也就是一旦有了第一个 gap,后面再接更多的 gap 就容易了,因为 gap 延 长罚分小。所以这种情况下,gap 都集中连成长串出现。
而反过来,当 gap 开头小,gap 延长大的时候,说明在连续的字母里插入一个 gap 打开一个缺口很容易,并不需要付出太大代价,因为 gap 开头罚分小。但是想在第一个 gap 后面再接一个 gap 就难了,因为 gap 延长罚分大。所以这种情况下很难有长串的 gap 出现,gap每延长一个都要付出巨大代价。因此在第二次我们做的结果里(图 2-A)都是分散的 gap。除了开头一段因两条序列长短不同而不得已出现的长串 gap 外,没有其他的长串 gap 了。

这就是说,通过调整 gap 开头和 gap 延长,我们可以把序列比对做成我们期待的样子。举两个例子,看看应该怎样调整 gap 最合理。第一个例子,你知道要比对的两条序列很相似,是同源序列,所以它们的结构和功能也应该都差不多。其中一条序列的结构已知,另一条未知。你想把它们很好的比对在一起,用其中已知结构的序列做模板,来预测另一个序列的结构。这时候我们期待得到的是 gap 分散的比对结果还是 gap 集中的呢?另一例子,你知道要比对的两条序列绝大部分区域都很相似,但是其中一条序列的一个功能区在另一条序列中是缺失的。你想要通过序列比对把这个功能区找出来。这时候我们要怎么设置 gap 开头和 gap延长呢?这两个例子告诉我们,在实际应用中,需要根据不同的情况选取不同的 gap 罚分,以满足不同的生物学意义。如果你对结果没有什么预期,那就请保持默认的参数。
其他在线双序列比对工具

| 软件名 | 比对类型 | 网址链接 |
|---|---|---|
| EMBL | Global/Local | http:/ / www. ebi.ac.uk/Tools/psa |
| PIR | Global | http://pir.georgetown.edu/pirwww/search/pairwise.shtml |
| Lalign | Global/Local | http://www.ch.embnet.org/software/LALIGNform.html |
| LAGAN | Global | http://agan.stanford.edu/laganweb/index.shtml |
| AligmMe | Alignment of Membrane Proteins | http://bioinfo.mpg.de/AlignMe/AlignMe.html |
| MCALIGN | Alignment fo non-coding DNA sequences | http://homepages.ed.ac.uk/eang33/mcalign/mcinstructions.html |
| Biotools | Global/Local | http://1.51.215.28/~gongj/biotools |
3.8 BLAST搜索
概念
我们已经学会如何做双序列比对,那可不可以拿一条序列和数据库中的每条序列逐一进行双序列比对,通过这种方法来找相似呢?这确实是一个办法。这样我们只要根据比对后得出的相似度排序,就可以找到最相似的那条序列了。但是,这种方法因计算耗时过长,只是理论上可行而已。之前我们用 EMBL 的双序列比对工具做全局比对,虽然很快就出结果了, 但至少也要经历一两秒钟的时间。而数据库中有几百万条序列,全部比对一遍,耗时太长。因此,我们需要快速的数据库相似性搜索工具。目前世界上广泛使用的就BLAST。它可以在尽可能准确的前提下,快速的从数据库中找到跟某一条序列相似的序列。
BLAST 是 Basic Local Alignment Search Tool 的首字母缩写,直译过来就是基本局部比对搜索工具。
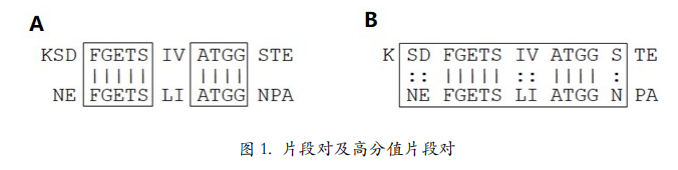
BLAST 的基本原理很简单,要点是片段对的概念。所谓片段对是指两个给定序列中的一对子序列,它们的长度相等,且可以形成无空位的完全匹配。(图 1-A )中方框里的就是两个片段对。BLAST 从头至尾将两条序列扫描一遍并找出所有片段对,并在允许的阈值范围内对片段对进行延伸,最终找出高分值片段对(high-scoring pairs, HSPs)(图 1-B)。这样的计算复杂度是 n 的一次方(n 是序列的长度)。如果做双序列比对话需要构建一个 n 乘以 n 的表格,计算复杂度是 n 的二次方。所以找高分值片段对比做双序列比对节省了大量的时间,当然,前提是牺牲了一定的准确度。
种类
(按搜索内容分类)
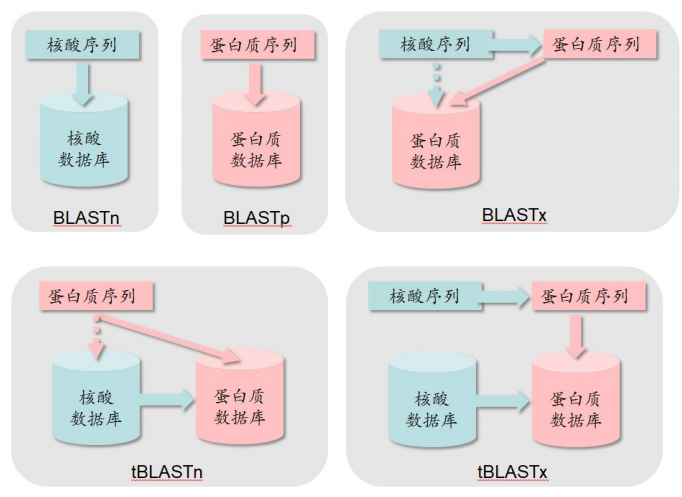
BLAST 实际上是综合在一起的一组工具的统称,它不仅可用于直接对蛋白质序列数据库和核酸序列数据库进行搜索,而且可以将待搜索的核酸序列翻译成蛋白质序列后再进行搜 索,或者反之,以提高搜索效率。因此 BLAST 可以分为 BLASTp,BLASTn,BLASTx,tBLASTn 和 tBLASTx。
- BLASTp 也就是用蛋白质序列搜索蛋白质序列数据库
- BLASTn 是用核酸序列搜索核酸序列数据库
- BLASTx 是将核酸序列按 6 条链翻译成蛋白质序列后搜索蛋白质序列数据库。
- 为什么是按 6 条链翻译?在无法得知翻译起始位点在情况下,翻译可能是从第1个碱基开始,三个三个的往后翻译,也可能是从第 2 个碱基开始,也可能从第 3 个碱基开始。另外还有可能是从 这条链的互补链上开始,这样又有3个可能的开始位置,加起来一共会产生 6 条可能被翻译出来的蛋白质序列。这 6 条中有些是真实存在的,有些是不存在,但是谁真谁假我们无从知晓,所以 6 条序列都要到数据库中去搜索一下试试。
- 既然是核酸序列,为什么不做 BLASTn 直接到核酸数据库里去搜索,而是要到蛋白质数据库里搜索呢?我们说这样做是有意义的,比如,从核酸序列数据库里找不到跟你手里这条核酸序列相似的序列,或 找到了相似的序列但这些找到的序列无法提供有意义的注释信息。这时,就可以去蛋白质数 据库试试,看看这条核酸序列的翻译产物能不能从蛋白质数据库里找到相似的序列以及有意 义的注释信息。或者说,你不是想找跟你这条核酸序列相似的核酸序列,而是想找跟你这条 核酸序列编码蛋白质相似的蛋白质序列,这时就要做 BLASTx。
- tBLASTn 是用蛋白质序列 搜核酸序列数据库,核酸数据库中的核酸序列要按 6 条链翻译成蛋白质序列后再被搜索
- 核酸数据库里不是已经注释了某条核酸序列能够翻译成什么蛋白质序列吗?为 什么还要把这些序列可能翻译出来的 6 条蛋白质序列都翻译出来搜索呢?我们说,你看到的 是已经注释的,还有没注释的呢!就算是已经注释的,你看到的也只是已经研究出来的成果, 还有没研究出来的呢!别忘了,基因可以重叠,注释上说某段 DNA 序列可以编码某个蛋白,但是可能某个未被发现的基因也用到了这段 DNA 序列。而你要搜索的这个蛋白质序列可能 刚好就是这个未被发现的基因的翻译产物。这样就必须把核酸序列所有可能的翻译产物都翻译出来,才能搜索得到。
- tBLASTx 是将核酸序列按 6 条链翻译成蛋白质序列后 搜索核酸序列数据库,核酸数据库中的所有核酸序列也要按 6 条链翻译成的蛋白质序列后再 被搜索。这样用 BLASTn 搜不着的,用 tBLASTx 就能搜着了。
BLASTp
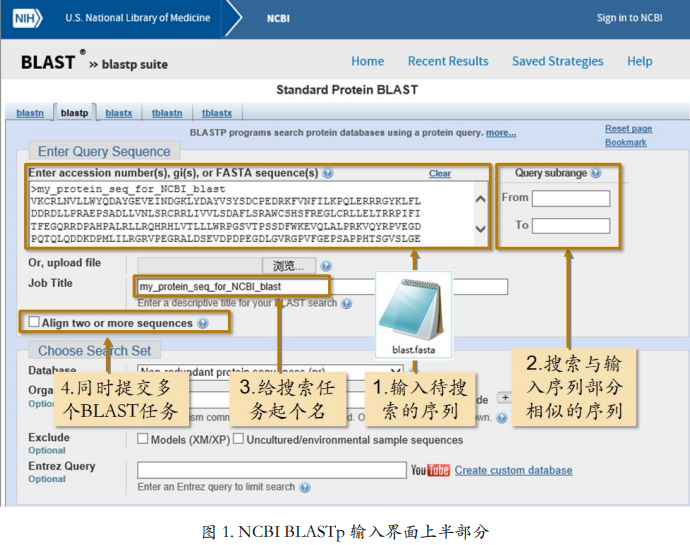
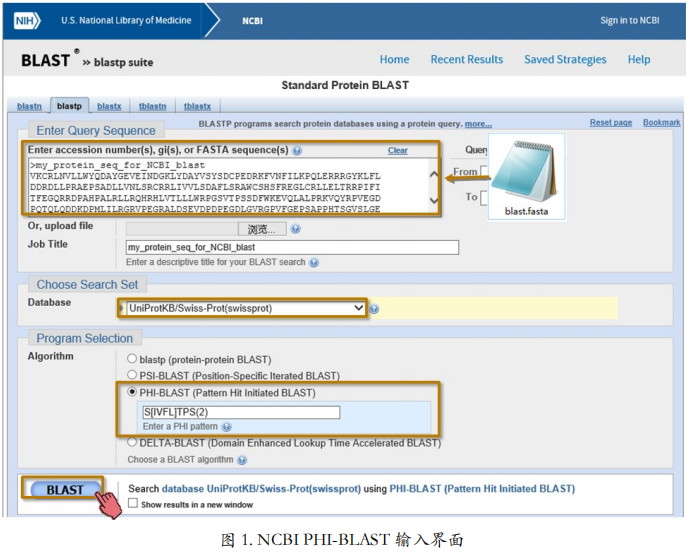
1> 在 BLASTp 输入界面里(图 1):
- 1)输入待搜索的蛋白质序列,这条序列可以在示例文件 blast.fasta 里面找到
- 2)指定搜索跟输入序列哪部分相似的序列,如果空着就是全长搜索
- 3)给搜索任务起一个名字,如果输入的是 FASTA 格式的序列,那么在输入框里面点一下,序列的名字就会被自动识别出来。
- 4)如果在 Align two or more sequences 前面打勾的话,可以同时提交多个 BLAST 任务。
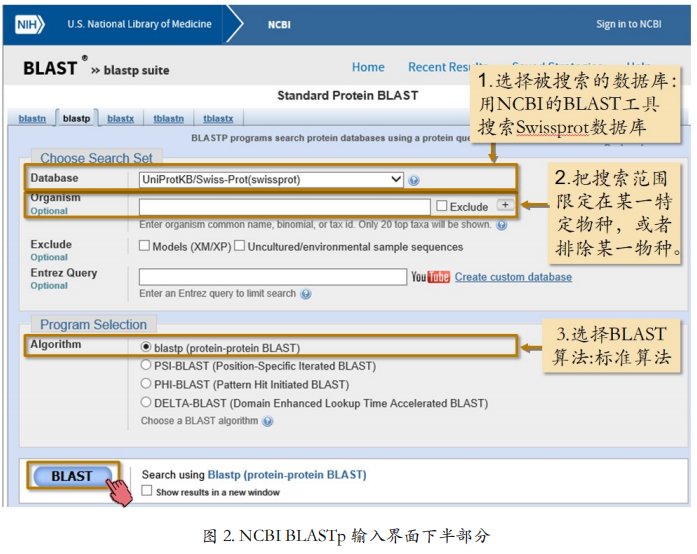
2> 在输入界面的下部(图 2)
- 1)选择被搜索的数据库
- 2)Organism 可以把搜索范围限定在某一特定物种内, 或者排除某一物种
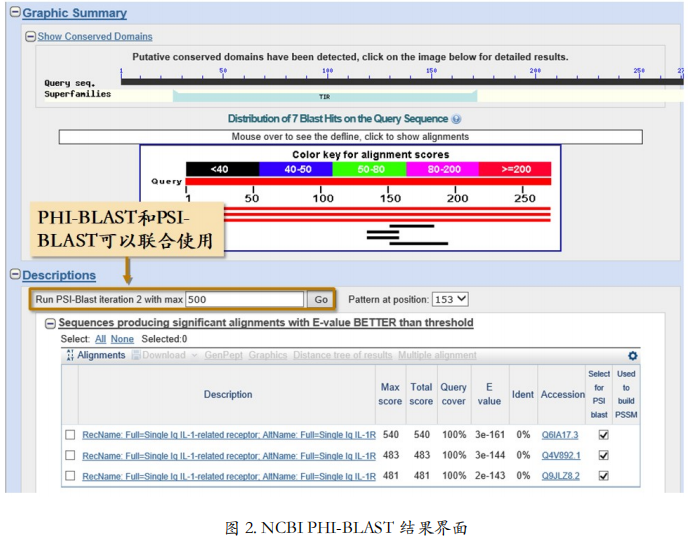
- 3)在算法选择这一栏里,有之前提到的三种不同的 BLAST 算法,标准BLAST,PSI-BLAST 和 PHI-BLAST
3> 图形化搜索结果部分
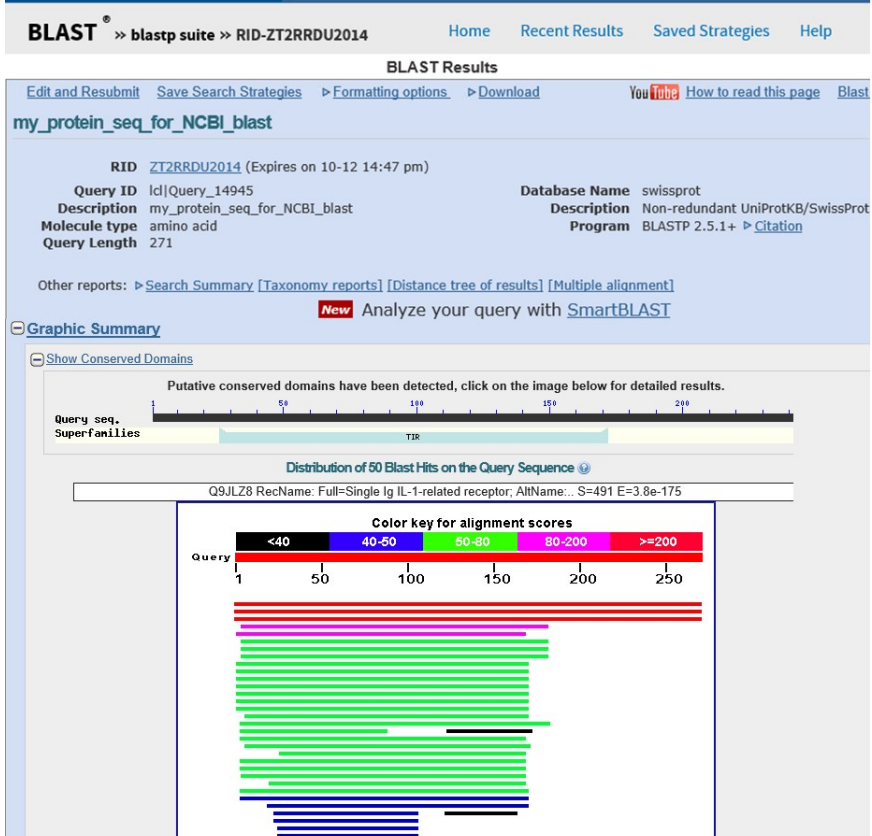
PSI-BLAST
为了提高速度,标准 BLAST 牺牲了一定的准确度,牺牲掉的准确度对高度相似的序列, 也就是亲缘关系近的序列构成不了威胁,不会把它们落掉,但是对于那些只有一点点相似, 也就是远源的序列,就有点麻烦了。它们很可能被落掉而没有被 BLAST 发现。
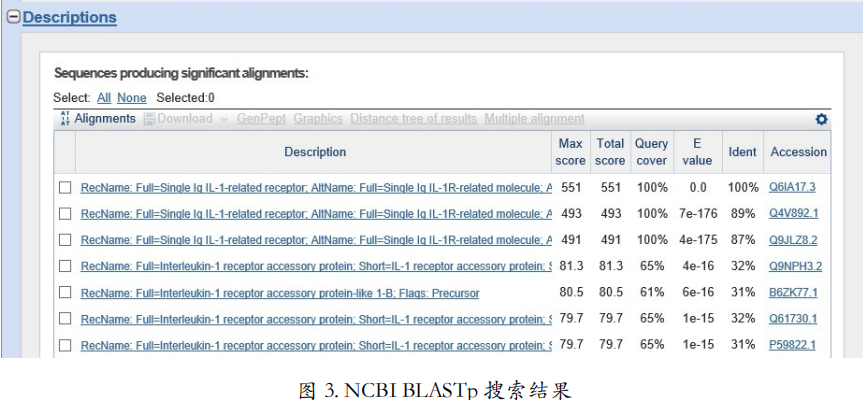
要解决这个问题,可以用 **PSI-BLAST**。PSI-BLAST 可以搜罗出一个庞大的蛋白质家族, 当然也包括标准 BLAST 不小心漏掉的那些远房亲戚。换言之,标准 BLAST 找到了直接认识的朋友,但朋友的朋友都丢掉了。PSI 是 **Position-Specific Iterated** 首字母缩写,中文是**位点特异性迭代**。
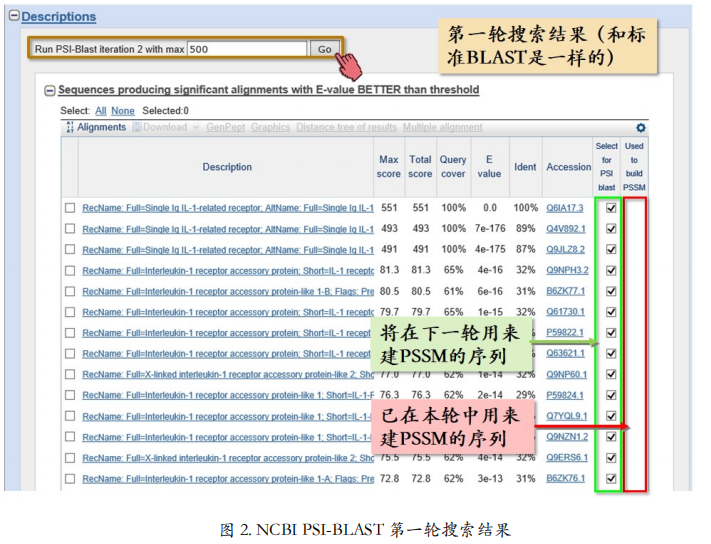
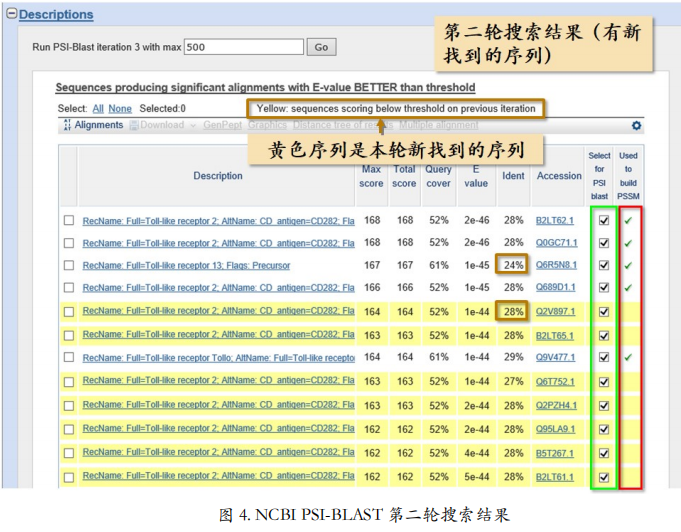
PSI-BLAST 的特色是搜完一遍再搜一遍,且从第二次搜索开始,每次搜索前都利用上一次搜索到的结果创建一个位置特异权重矩阵以扩大本次搜索的范围。如此反复直至没有新的结果产生为止。位置特异权重矩阵(Position-Specific Scoring Matrix,简称 PSSM)是 以矩阵的形式,统计一个多序列比对中,每个位置上不同残基出现的百分比。假设 A 的朋 友只有 B,B 朋友除 A 外还有 C。如果输入序列的第一个位置是 A,那么在第一轮没有 PSSM 辅助的情况下,只有第一个位置是 A 或 B 的序列被找到了。它们是图 1-A 中所示的四条序列。根据这四条序列创建的 PSSM(图 1-B)得知,第一个位置可以是 A,也可以是 B,那么在第二轮搜索中,除了 A 的朋友 B 之外,B 的朋友 C 也可以出现在第一个位置了。这样如此反复,我们就可以找到朋友的朋友了。

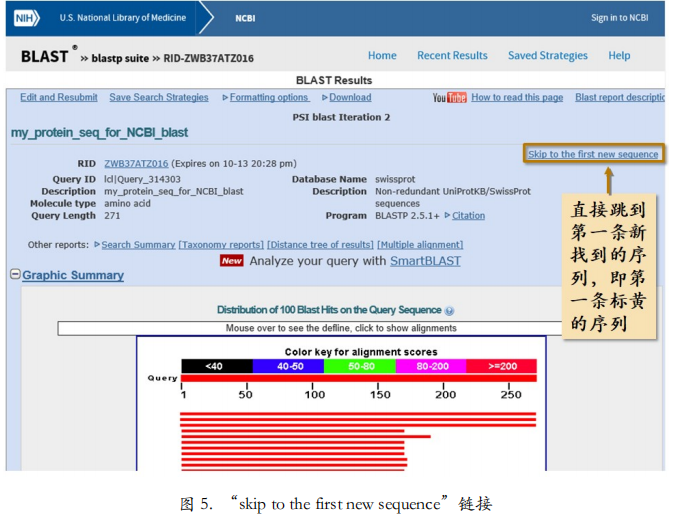
PHI-BLAST
PHI-BLAST 和 PSI-BLAST 不同,PSI-BLAST 是撒大网搜索,而 PHI-BLAST 则是精准搜索。PHI 是 Pattern-Hit Initiated 首字母缩写,中文是模式识别。PHI-BLAST 能找到与输入序列相似的并符合某种特征模式的蛋白质序列。模式 Pattern 是对特征的描述。
特定模式可通过正则表达式来表述。所谓正则表达式就是这句话的一个简约的规范性字 符书写法。发生 N 糖基化位点的序列符合的特定模式翻译成正则表达式为 N{P}[ST]{P}。 其中,N 是天冬酰胺,P 是脯氨酸,S 是丝氨酸,T 是苏氨酸。{}代表除大括号里的氨基酸 以外的任意氨基酸,[]代表中括号中的任意一个氨基酸。得知这些符号的含义之后,这个 正则表达式就很容易读懂了。PHI-BLAST 可以根据给入的正则表达式对搜索到的相似序列进行模式匹配,符合正则表达式的才会被作为结果输出。
熟悉一下正则表达式,{}代表除什么以外,[]代表其中之一,x 代表任意字 母,(3,7)代表 3 到 7 个某字符。那么正则表达式{L}GEx[GAS][LIVM]x(3,7)的意思是, 除 L 以外的任意一个字母,紧接 G,再紧接 E,再接一个任意字符,之后是 GAS 中的任意 一个,再接 LIVM 中的任意一个,最后再接 3 到 7 个任意字符。
在 NCBI BLAST 工具的输入页面,当算法选择了 PHI-BLAST 之后,会自动出现模式输 入框(图 1)。输入正则表达式 S[IVFL]TPS(2)(含义为:一个 S 后面紧接 IVFL 中的任意 一个字母,再接 T,再接 P,再接两个 S)。这次搜索找到的相似序列中,只有符合该模式的才会被作为结果返回。
SMART-BLAST
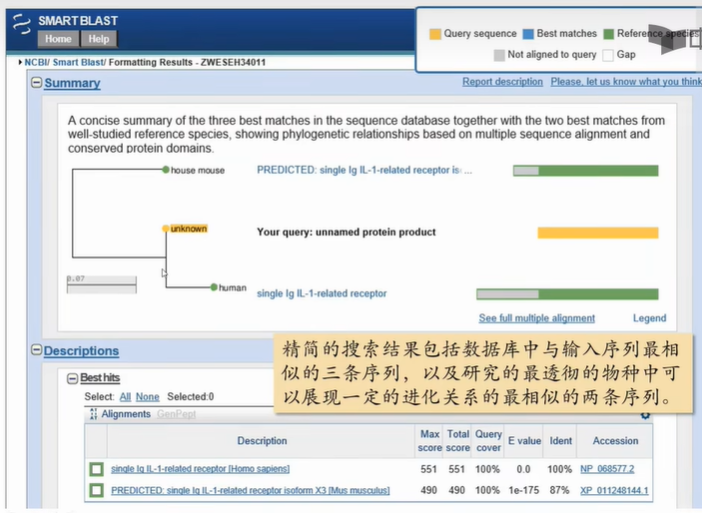

| 位置 | 服务器 | 网址链接 |
|---|---|---|
| USA | NCBI | http://www.ncbi.nlm.nih.gov/BLAST |
| Europe | ExPASys | http://web.expasy.org/blast |
| Europe | Uniprot | http://www.uniprot.org/blast/ |
| Japan | DDBJ | http://blast.ddbj.nig.ac.jp |
3.9 多序列比对介绍
用途和算法
用途
- (1)我们可以通过多序列比对确定某一个未知序列是否属于某一个家族。
- (2)可以用多序列比对构建系统发生树,查看物种间或者序列间的进化关系。事实上, 做多序列比对是构建系统发生树的必要步骤之一。
- (3)模式识别。一些特别保守的序列片段往往对应着重要的功能区。通过多序列比对,可以找到这些保守片段,并由此推测出潜在功能区。
- (4)可以把已知的有特殊功能的序列片段通过多序列比对做出匹配模型。然后根据这个模型推测未知的序列片段是否也具有这个功能
- (5)除此之外,多序列比对在生物信息学分析的很多方面都有应用,比如用来预测蛋白质的二级结构和三级结构,预测 RNA 的二级结构等等。由
算法
两条序列的比对需要构建一个二维表格,然后从 右下角到左上角找出一条最优路线。
如果是做 3 条序列的比对,应该做一个三维立方体,从 (0,0,0)这个位置到(n,n,n)这个位置找到最优的贯穿路径。
以此类推,如果是做 n 条序列的比对,就要创建一个 n 维空间。这个 n 维空间实在是难以想象,但是有一点是明确的, 就是到了 n 维我们已经没有办法再像二维那样精确的计算出比对结果了。
由于计算量过于巨大,所以目前所有的多序列比对工具都是不完美的。它们都使用一种近似的算法。目的就是为了缩短计算时间,但也因此牺牲了一定的准确度。好在多序列比对并不像双序列比对对准确度要求极高。通常,我们是要从多序列比对中看到一个趋势,一个大体的位置,所以牺牲掉的这点儿准确度影响不大。
注意事项
序列选取
- (1)做多序列比对的序列个数不能太多,一般 10 到 15 条序列刚好,最好不要超过 50 条。序列太多,任何软件都受不了。
- (2)关系太远的序列不适合做多序列比对。两两之间序列相似度低于 30%的一组序列, 做多序列比对要么做不出来,要么即使勉强做出来了,做得也是零七八碎,没有任何意义。
- (3)关系太近的序列不适合做多序列比对。两两之间序列相似度大于 90%的序列,有再多条都只等于一条。做出来的多序列比对无非就是把各条序列抄写了一遍,没有任何意义。
- (4)短序列受不了。多序列比对支持一组差不多长的序列,个别很短的序列纯属捣乱分子,受不了。
- (5)有重复域的序列受不了。如果序列里包含重复片段,大多数多序列比对的程序都会出错,甚至崩溃。
序列起名
(1)序列名字里不要有“空格”,用下划线代替“空格”是个好习惯。
(2)不要使用特殊字符,比如中文,@,#之类的。
(3)序列名字不要太长,最好在 15 个字符以内。
(4)一组序列里,不要有重名的序列。
如果不按上述几点建议命名的话,多序列比对工具会在不告诉你的情况下修改你的序列名称。比如这个名字“This_is_my_favorite_sequence_about_mouse”是一定会被程序截短的。通常只保留前 15 个字符。如果区分各个序列的关键词恰好在后面,那么截短之后,所有序列的名字都一样了,很难分辨。掌握了这几点之后就可以开始做多序列比对了。
3.10 在线多序列比对工具
目前世界上最流行的多序列比对工具是 CLUSTAL 系列,TCOFFEE 和 MUSCLE。其中 CLUSTAL 系列使用率最高;TCOFFEE 最新,而且还有很多变形;MUSCLE 最快,而且胃口大,能接受的序列数量是其他工具比不了的。各大生物网站,比如 EBI、Expasy 等都可以 在线使用这些工具。当然它们也有自己的网站。
EMBL-Clustal Omega
我们先介绍EMBL的多序列比对工具。EMBL 的多序列比对工具很多,包括前面提到的 CLUSTAL 系列、TCOFFEE、MUSCLE。我们看 EMBL这些比对工具中 CLUSTAL 系列的最新版本 Clustal Omega。
输入界面
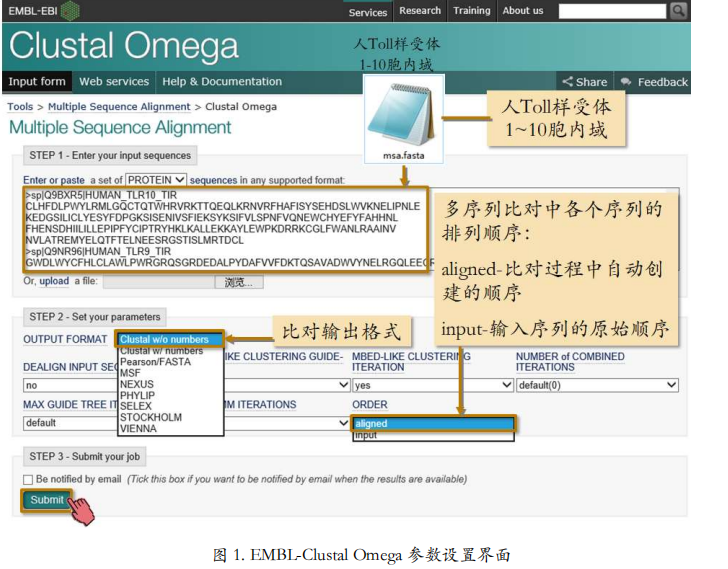
要比较的多条序列存放在 msa.fasta 文件中。这个文件里存了 10 条序列,每条序列都用FASTA 格式书写。这样程序会根据 “>” 自动识别出每条序列以及它们的名字。这就是 FASTA格式的好处。进入Clustal Omega参数输入界面(图 1),黏贴或者上传序列。
参数设置
在这个界面里可以点通过点击 More options,设置各种设置。在这个例子里,所有参数都使用默认值。参数里有输出格式(OUTPUT FORMAT)和输出顺序(ORDER)这两个参数。
输出格式里可以选择常用的多序列比对格式。我们选标准的 Clustal 格式。这是最常见的多序列比对格式。
输出顺序参数可以设定多序列比对中各个序列的排列顺序。“aligned”是按照比对过程中自动创建的计算顺序排列;“input”是按照输入序列的原始顺序排列。
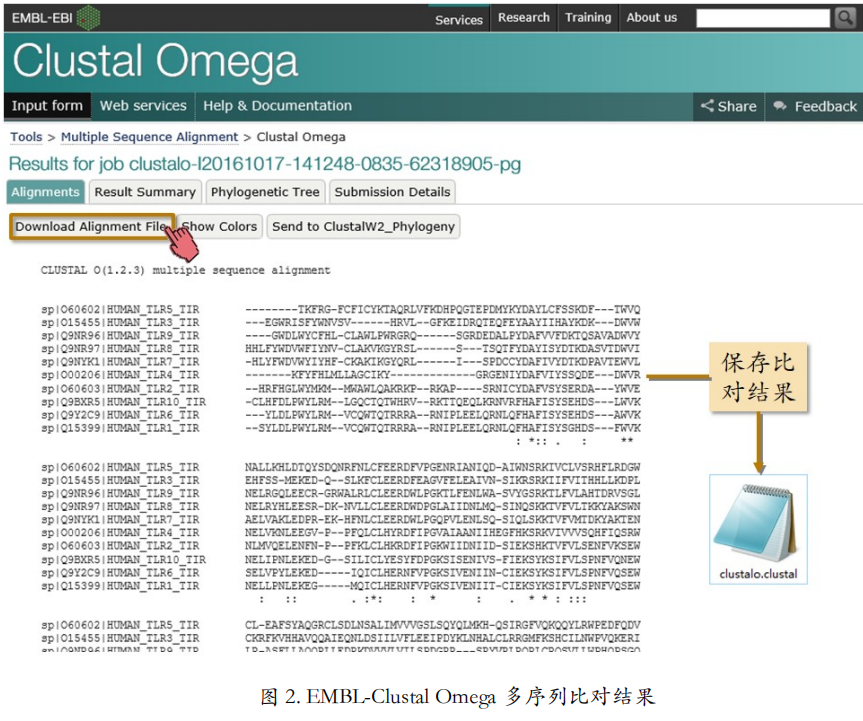
比对结果
做多序列比对的时间要比双序列略长。序列越多,序列越长,则时间越长。Clustal 格式的输出结果如图 2 所示。可以看到,比对中序列的排列顺序跟输入的时候不一样了,这是按 照比对创建时的计算顺序排列的。请点击 Download Alignment File 保存将当前结果,以便后 面章节进一步加工。保存的文件后缀名是“.clustal”。它是一个纯文本文件,用写字板或者记事本都可以打开
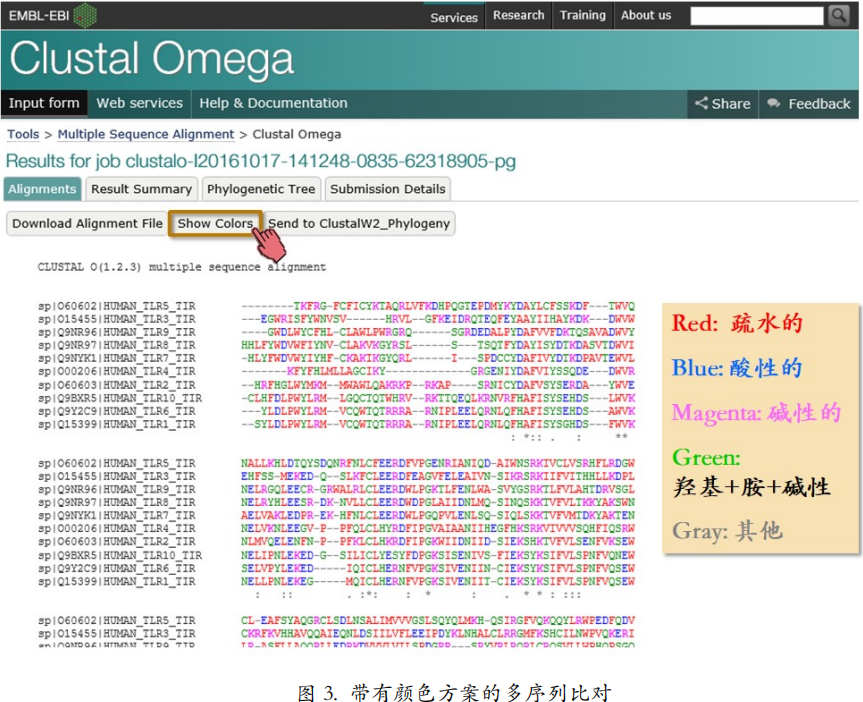
如果想要添加色彩,点击“Show Colors”。之后,不同的氨基酸根据它们的理化性质 不同会显示出不同的颜色(图 3)。
除了颜色之外,多序列比对每一段的最后一行都有些星星点点的标记。这些标记和双序列比对中的竖线、双点、单点的意思类似,但并不完全相同。
如果某一列是完全保守的一列,也就是说这一列里的字母完全相同,那么这一列的下面就打一个“*”。
如果这一列的残基有大致相似的分子大小及相同的亲疏水性,也就是这一列的字母要么相同要么相似,没有不相似的,那么就打一个“:”。
如果这一列残基的分子大小及亲疏水性被一定程度上保留了,但是有替换发生在不相似的残基间,也就是这一列的字母有相似的也有不相似的,那么就打一个“.”。
什么都不标记代表这一列是完全不保守的,也就是这一列的字母全部都不相似。
这些星星点点的标记可以为我们指出保守区域所在的位置,即,星星点点特别密集的区域。
结果总结
Result Summary标签里,给出了全部结果信息的下载列表和一个 Jalview 的按钮(图 4)。
Jalview 是多序列比对编辑软件,之后的章节里面我们会详细介绍。
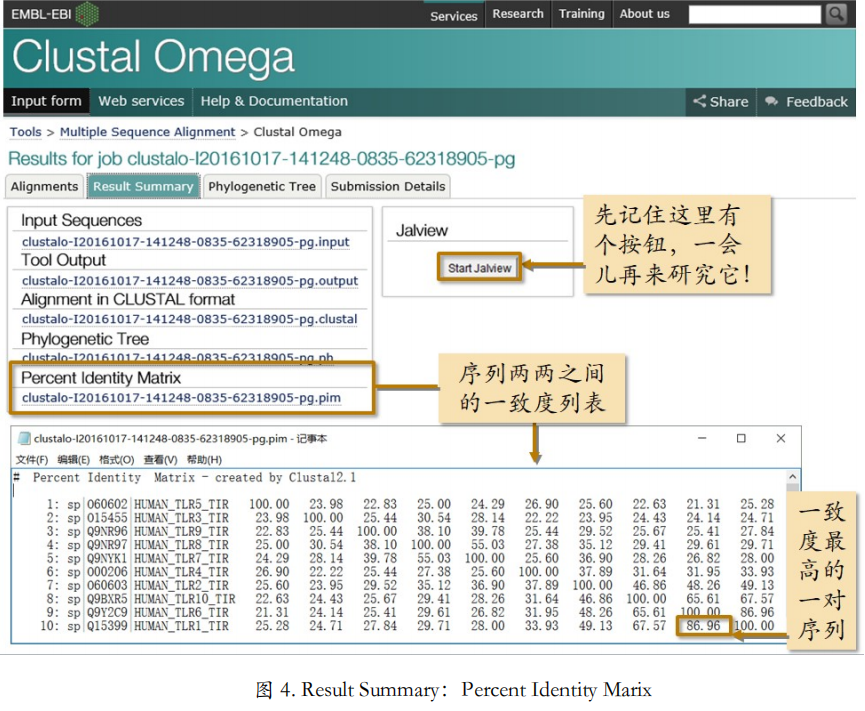
在下载列表里,如果打开“Percent Identity Matrix”链接,可以得到所有序列两两之间的一致度矩阵。一致度矩阵的第一行省略掉了。它和第一列完全相同,都是序列的名字并且按照相同的顺序排列。所以这个矩阵是以对角线为轴对称的,并且对角线上是某条序列自己和自己的一致度,都是 100%。这个矩阵可以帮助我们更好的了解这些序列之间的关系。 比如我们可以从中发现,一致度最 高的一对序列是 TLR1 和 TLR6。
系统发生树
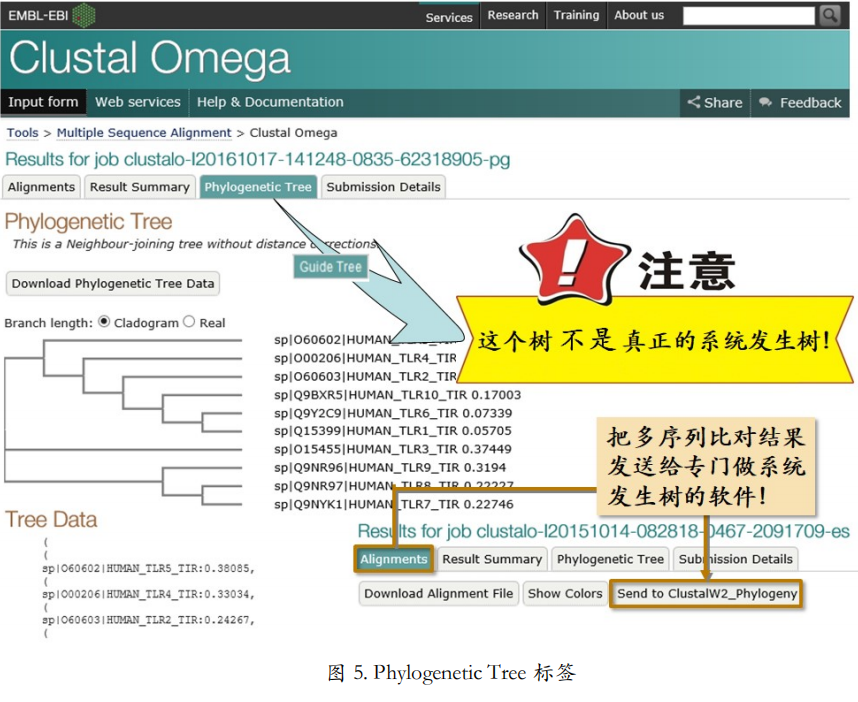
除了通过一致度矩阵了解序列间的关系,还可以通过 Phylogenetic Tree 标签下的 Guide Tree 清楚的看出哪条序列和哪条序列更相似(图 5)。Phylogenetic Tree 翻译成中文是系统发生树。但是这里要特别注意,这不是真正意义上的系统发生树!它只是在创建多序列比对的过程中用到的树(Guide Tree),没有经过距离校正,所以不能当作系统发生树来使用。如果 想要根据多序列比对结果构建系统发生树,可以在 Alignments 标签下,点击“Send to ClustalW2_Phylogeny”链接,把做好的多序列比对发送给专门做系统发生树的工具。
TCOFFEE-Expresso
TCOFFEE 是一个非常流行的多序列比对工具。TCOFFEE 与 CLUSTAL 系列在所使用的算法上类似,准确度上比 CLUSTAL 系列略高,但计算耗时也比 CLUSTAL 系列略高。最关键的是 TCOFFEE 有很多种变形,也就是说它有更多的功能。许多网站都提供 TCOFFEE的在线使用,比如 EMBL 的多序列比对工具里就有 TCOFFEE。但是这次,我们从 TCOFFEE的网站做多序列比对。
比对工具
TCOFFEE 本身是一个标准的多序列比对工具,跟 CLUSTAL 没有什么区别。我们来看 它的变形,也就是根据比对序列种类的不同,TCOFFEE 网站下特有的比对工具(图 1)
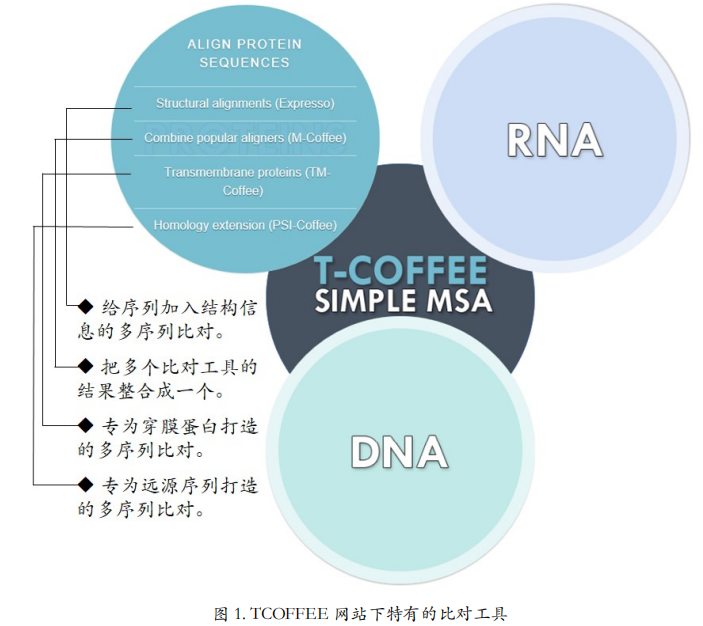
针对蛋白质序列的比对工具,除了 TCOFFEE 以外,还有 Expresso,M-Coffee, TM-Coffee以及 PSI-Coffee。其中,
- Expresso 最有特色,它是为序列加入结构信息后再做多序列比对的工具。因为有结构信息的辅助,它可以大大提高比对的准确度。
- M-Coffee 可以把多个比对的结果整合成一个。
- TM-Coffee 专为穿膜蛋白打造,
- PSI-Coffee 专为远源序列打造。
- 同样的还有针对 RNA 和 DNA 序列的 Coffee。
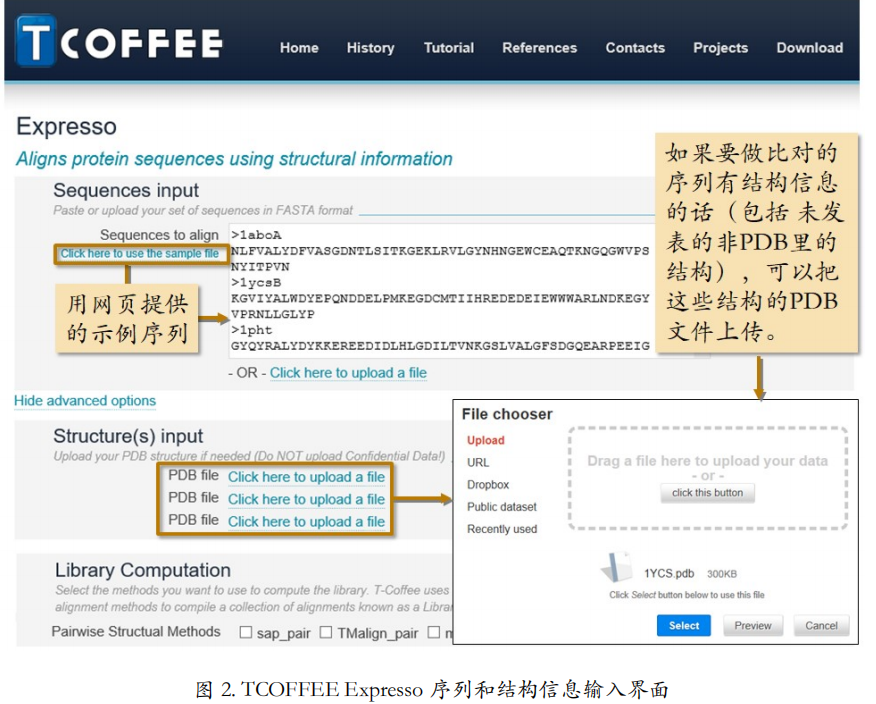
Expresso输入界面
做 Expresso 的序列我们选用网站提供的示例序列(图 2)。Show more options 下,可以通过各种方式给入输入序列的结构信息。如果你有这些序列现成的结构文件,也就是 PDB文件,可以直接把它们上传上来。三条序列对应三个上传链接。可以上传的结构文件不只限于 PDB 数据库下载的,也包括还未正式发表的解析结构或者计算机预测的结构,只要是用PDB 文件格式保存的,都可以。
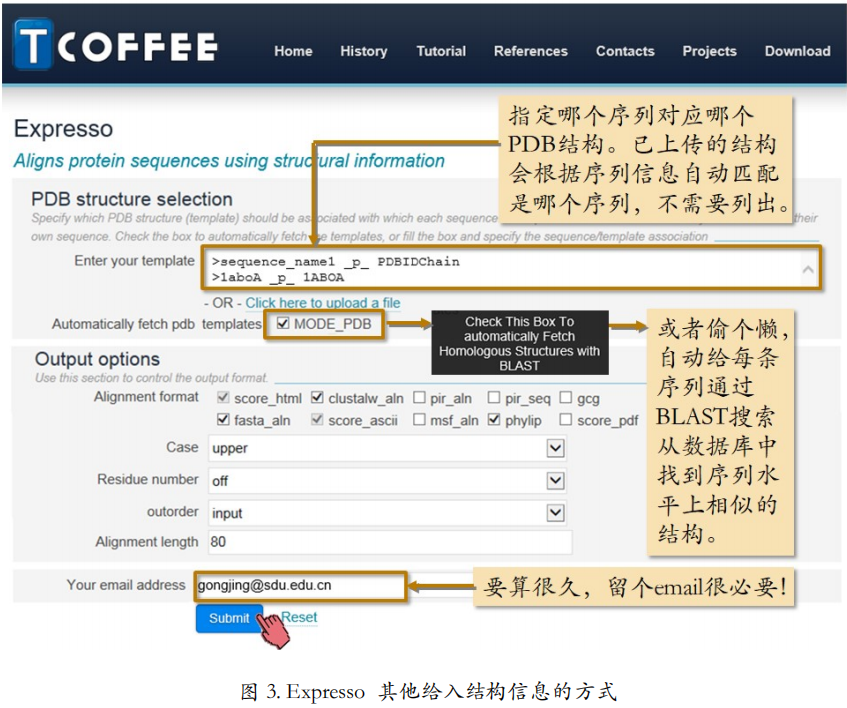
如果没有现成的结构文件,但是这些序列在 PDB 数据库里有对应结构的话,你可以从接下来的输入框里,按照规定的写法,指定哪条序列对应 PDB 数据库中的哪个结构(图 3)。 如果这里输入了信息,Expresso 会自动从 PDB 网站下载指定的 PDB 文件。
那些已经本地上传的结构,Expresso 也会根据序列信息自动匹配出它是哪条序列的结构,不需要再在这里列出了。
如果对结构信息一无所知,只需要将“MODE_PDB”钩选。之后,Expresso 就会自己在网络上为所有没有指定结构信息的序列搜索相应的结构。你提供给 Expresso 的结构信息越多,计算时间就会越短;你提供结构信息越少,计算时间就会越长。如果只勾选了 “MODE_PDB”,那么需要等待的时间会很长,因为 Expresso 首先要搜索,然后要下载, 最后要计算。因此,留下你的 Email 信息是很有必要的。
比对结果
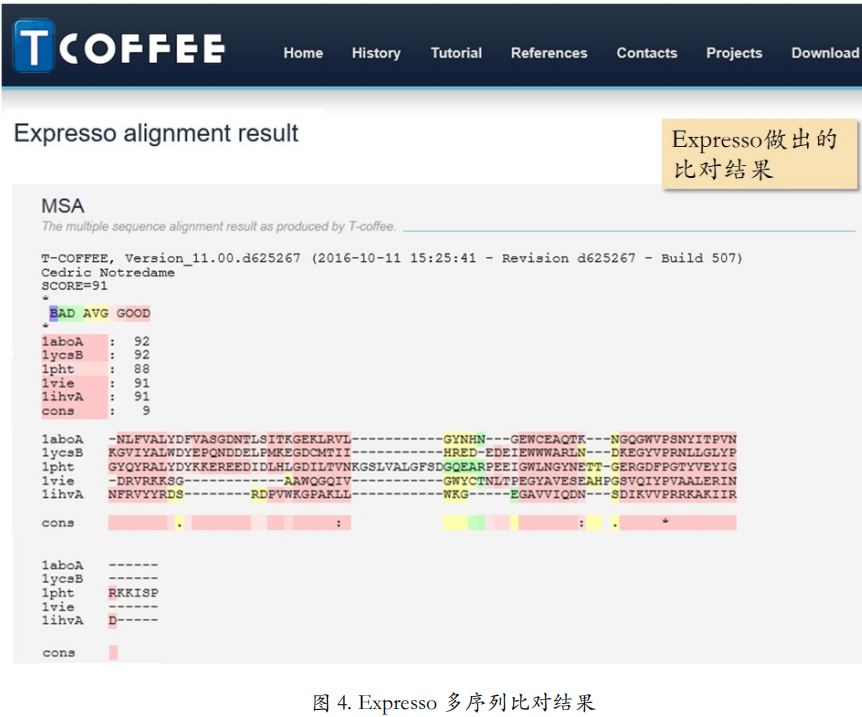
比对结果的页面链接会发送至邮箱。打开链接后会得到 Expresso 做出的比对结果(图 4)。 TCOFFEE 系列各比对工具做出的多序列比对的颜色代表比对质量的好坏。越红质量越好, 越蓝质量越坏。这次的比对结果非常令人满意。如果配合上这些序列二级结构信息看一下的话,你会发现,螺旋和螺旋很好的对在了一起,折叠和折叠很好的对在了一起。
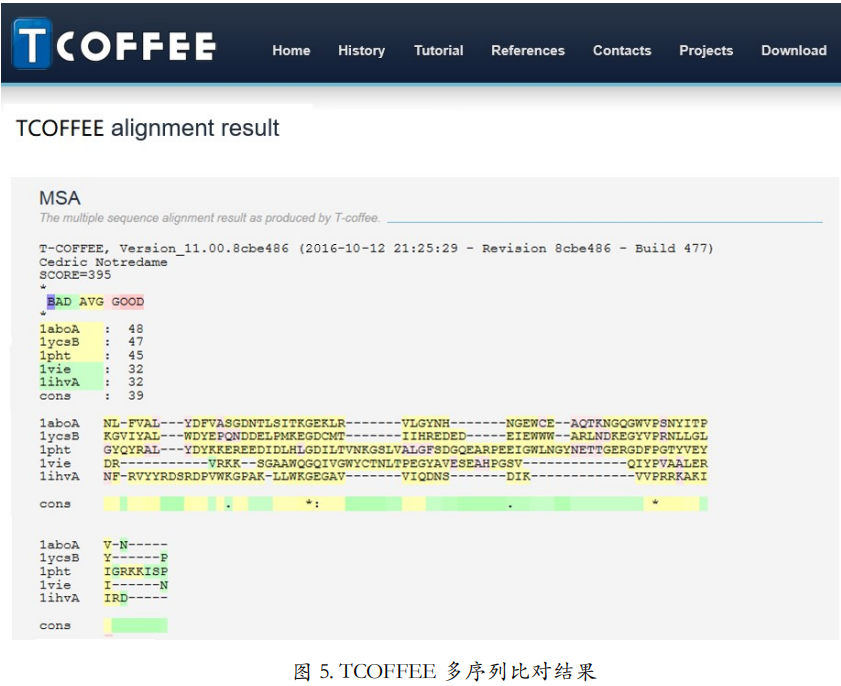
同样的序列做普通的 TCOFFEE,质量远不如 Expresso(图 5)。可以看到二级结构全部错位。所以,如果你有序列的结构信息的话,用 Expresso 相比用普通的比对工具会大大提高比对质量。
多序列比对的保存格式
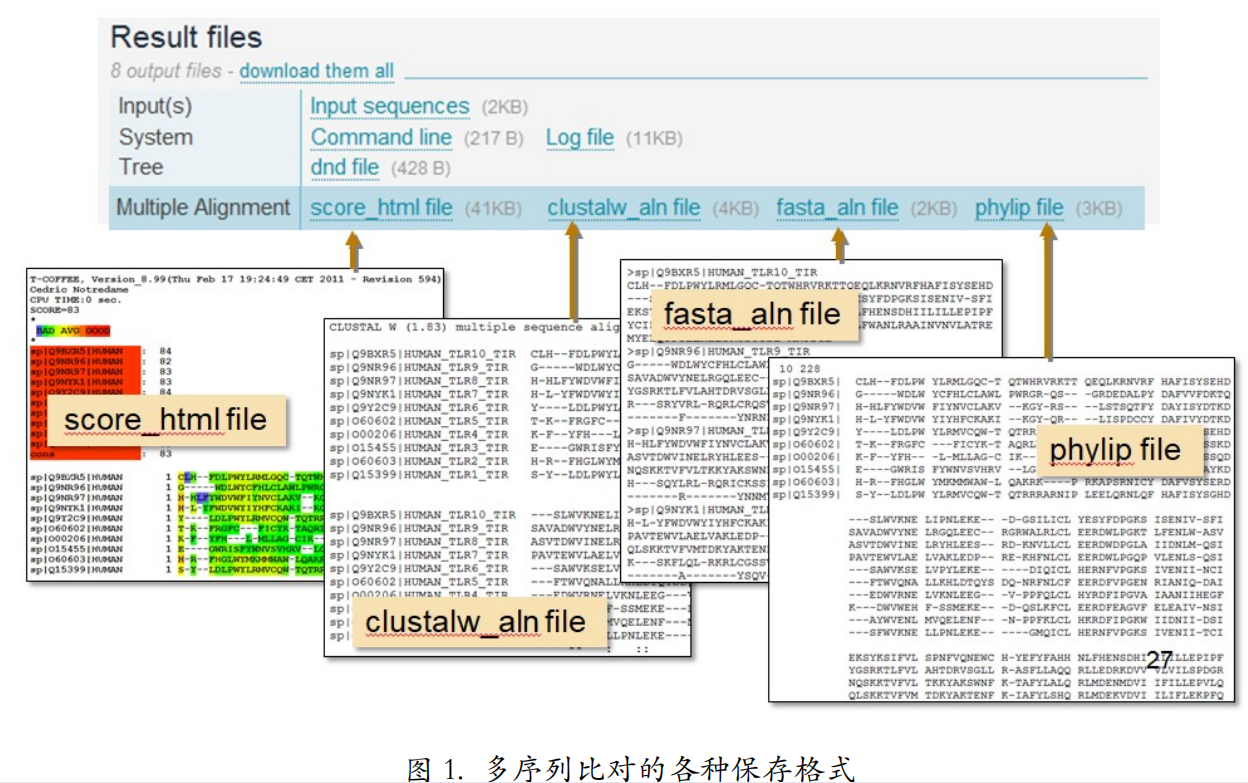
无论用哪种多序列比对工具,CLUSTAL 系列也好,TCOFFEE 系列也罢,都提供多种格式的多序列比对结果。比如有漂亮的网页格式的,标准的 Clustal 格式的,还有写完一条 序列再写下一条的 FASTA 格式的,以及方便下一步建树使用的 Phylip 格式的(图 1)。那到 底保存哪种格式好呢?
要保存哪种格式主要看你下一步要干什么。在选择保存格式之前,需要问自己几个问题。
1)你选的这个格式大多数软件都支持吗?
2)你的同事能用吗?
3)你需要的信息这个格式都提供了吗?
4)这个格式适合进一步加工吗?
通过回答这几个问题来确定你最终需要的格式。如果比对工具输出的格式里没有你想要的哪种,可以通过第三方软件进行格式转换。比如 fmtseq工具,它可以实现 20 多种格式间的转换。其中总有一款是你想要的。
3.11 多序列比对的编辑和发布
Jalview 的介绍
比对工具刚刚做出来的多序列比对有点儿生,我们通常还需要给它们加工一下。这就需要多序列比对结果编辑器。它能给比对结果添加各种颜色,还能手动编辑比对。常用的比如 Jalview。这个名字我们之间见过。在 EMBL Clustal Omega 比对结果的 Result Summary 标签下有 Jalview 按钮(图 1)。这个按钮可以快速启动 Jalview,但这里启动的在线版本功能不完整。
完全版的 jalview 可以从 Jalview 官网在线启动,或者下载安装到本地。从这个图 2 可以看出,Desktop 版的 Jalview 和许多序列分析软件及生物数据库之间都有关联。通过 Jalview 除了可以加工多序列比对,还可以针对比对中的序列做各种各样的分析、比如构建系统发生树、预测蛋白质二级结构、查看结构域家族、从 PDB 数据库中查询三级结构等。此外 Applet 版的 Jalview 可以被加载到网页中,就像 EMBL 的多序列比对工具那样,可以从网页上直接运行并显示某个比对结果
在线启动 Desktop 版的 Jalview 可以点击主页右上角的“Launch Jalview Desktop”链接。Jalview 基于 java 编写,所以要安装 java、信任 java、接受 java、运行 java。主页上在线打开的Jalview 也并没有安装到本地,只是下载运行了一个远程连接的小插件而已。断网会导致Jalview 关闭。但这个版本的 Jalview 的功能是完整的。
如果想要将 jalview 安装到本地,获得更稳定的使用体验,可以点击主页上的“Download”链接。目前 Jalview 支持几乎所有的操作系统,选择适合你的安装。此外,Windows 版本的安装文件也可以从附件里面下载。

Jalview 的操作
导入文件
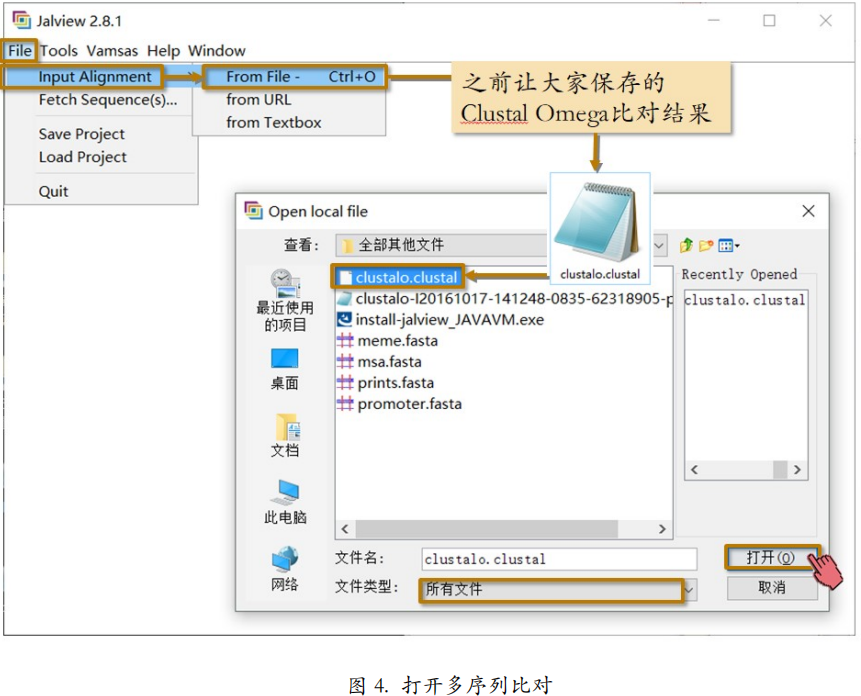
Jalview 打开之后,会自动展示许多 Demo 窗口(图 3)。通过这些窗口,你可以了解到,Jalview 能加工序列比对、做进化树、分析结构,等等。功能确实不少。不过,你需要做的,是把这些窗口统统关掉。打开你自己的多序列比对(图 4)。点击 File 菜单 - Input Alignment - From File - 打开我们之前用Clustal Omega做出并保存的多序列比对结果“clustalo.clustal”(如果你忘记保存了可以从附件中下载)。因为“.clustal”不是 Jalview 熟悉的后缀名,所以需要把文件类型改成“所有文件”才能看到它。
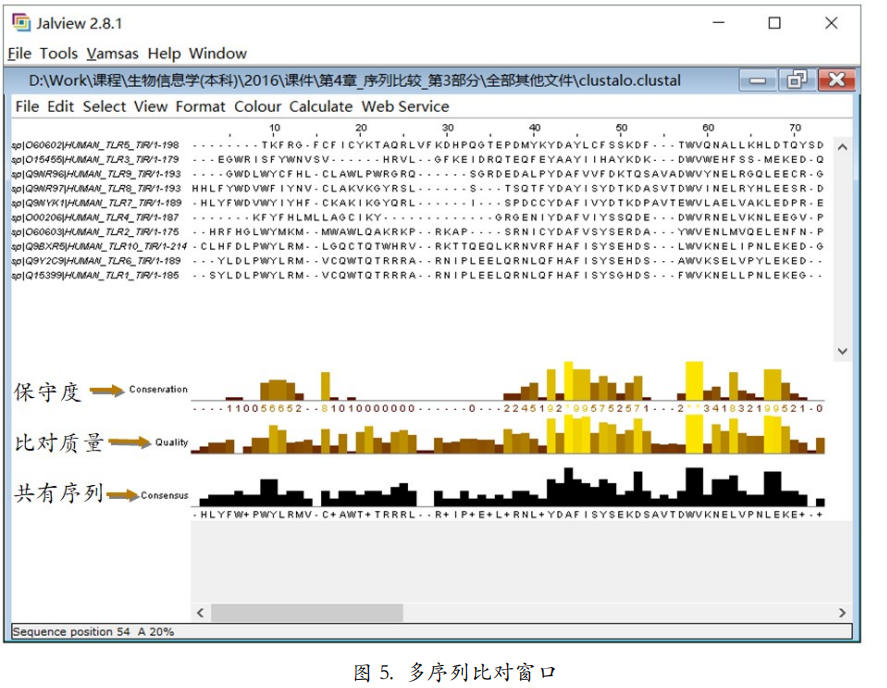
在打开的多序列比对窗口的下方有三行柱状图(图 5)。它们体现了比对中每个位置的保守度高低(Conservation)、比对质量高低(Score)、以及共有序列(Consensus)。
- 从保守度行,可以很清楚的找到保守区大致的位置。
- 共有序列指的是某一列出现频率最高的那个字母, 比如第 58 列中 W 出现的频率最高,是 100%。如果某一列拥有的最高出现频率的字母是两或两个以上的话,会以“+”显示。把鼠标放在“+”上就可以看到是哪些字母出现的频率一样高。共有序列可以一定程度上体现出某个保守区域所具有的序列特征。以后如果看到和这段序列长相极其相似的序列,它很可能跟这个保守区的功能相似。
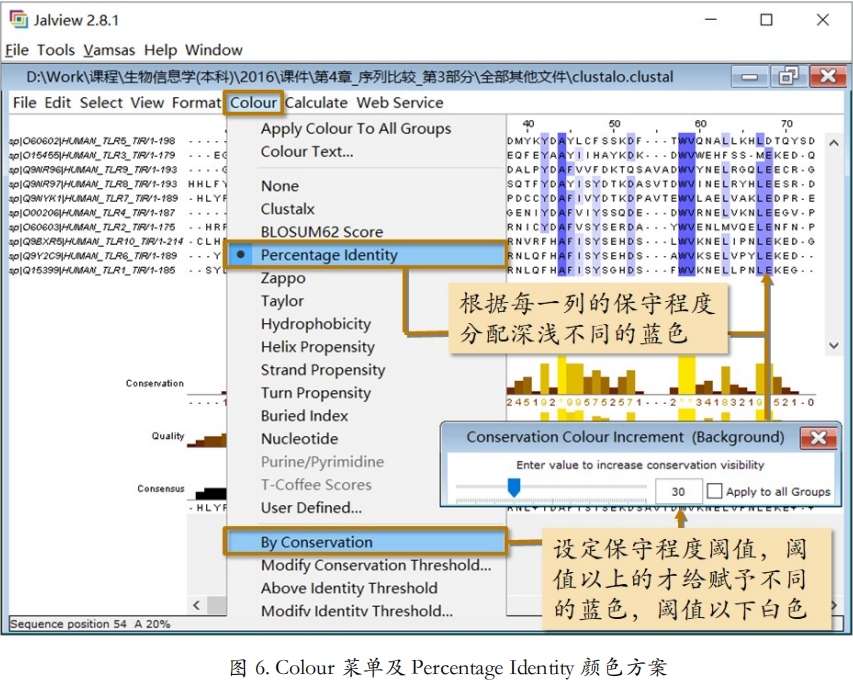
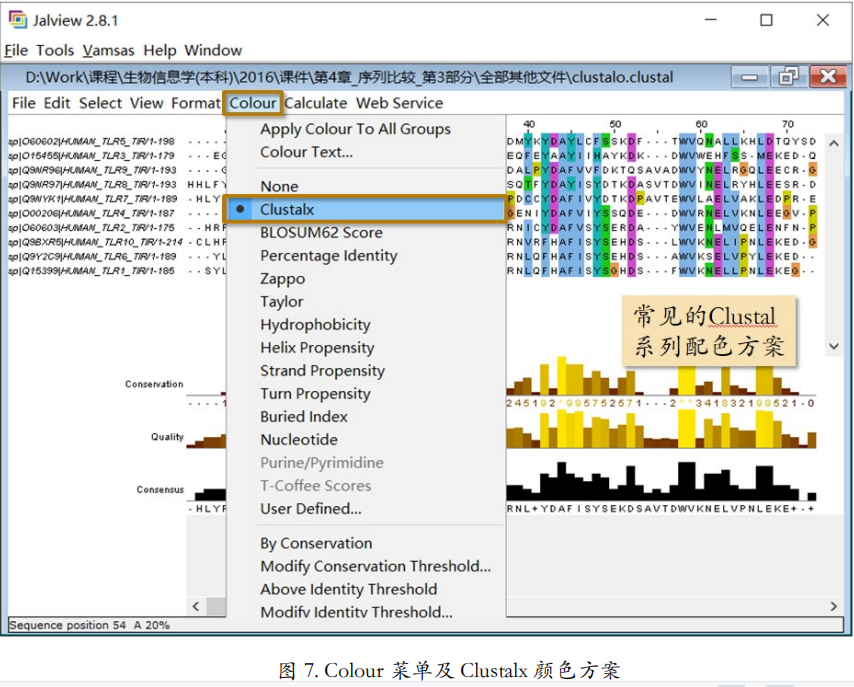
序列上色
现在我们开始加工序列比对。首先先上颜色,在Colour 菜单下有很多种颜色方案(图 6)。能够和保守度这一行柱状图配合的颜色方案是 Percentage Identity。选了这个颜色方案之后, 每一列会根据这一列的保守度用深浅不同的蓝色表示。蓝色越深说明这一列越保守,反之越不保守。再配合 Colour 菜单下的“By Conservation”参数,可以从弹出的参数设定窗口中设定保守程度达到百分之多少以上的才给赋予不同的蓝色,阈值以下的都是白色。
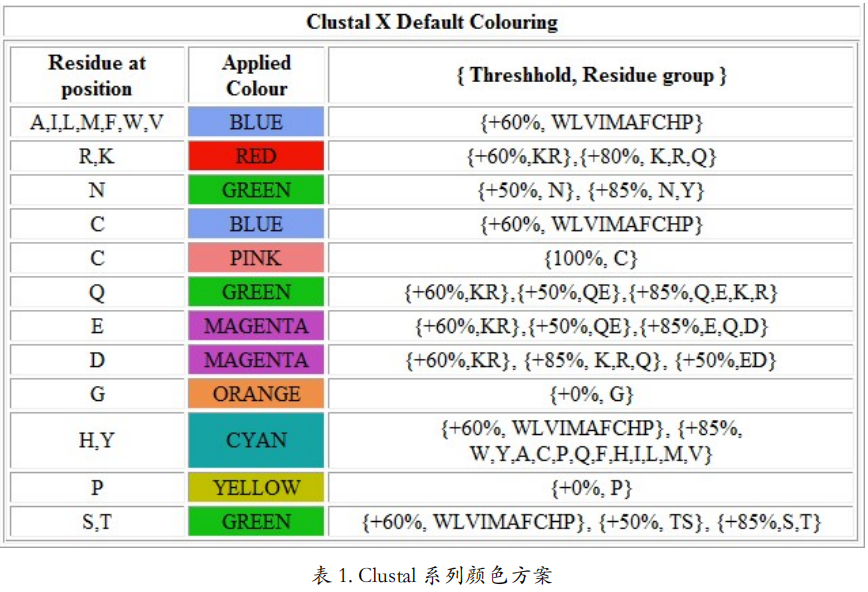
另一个较常用的颜色方案是 Clustal 系列配色方案。这个配色方案和 EMBL 多序列比对工具做出的结果页面里“Show Colors”之后的颜色方案是基本相同的。具体哪个氨基酸选用哪个颜色可以参见表 1。我们从文献里看到的彩色多序列比对,大多是用的这种颜色方案。
序列修瑕
除了给多序列比对上彩妆,有时还需要给它修理一下局部瑕疵,也就是对局部位置进行手动调整。比如,从前期实验我们得知,图 1 中方框所示区域的 TLR2、10、6、1 这四条序 列的第 53 列应该往右挪一列,跟 TLR9、8、7 这三条序列的第 54 列对在一起。TLR2、10、6、1 这四条序列的第 53 列补空位。其他位置不动。
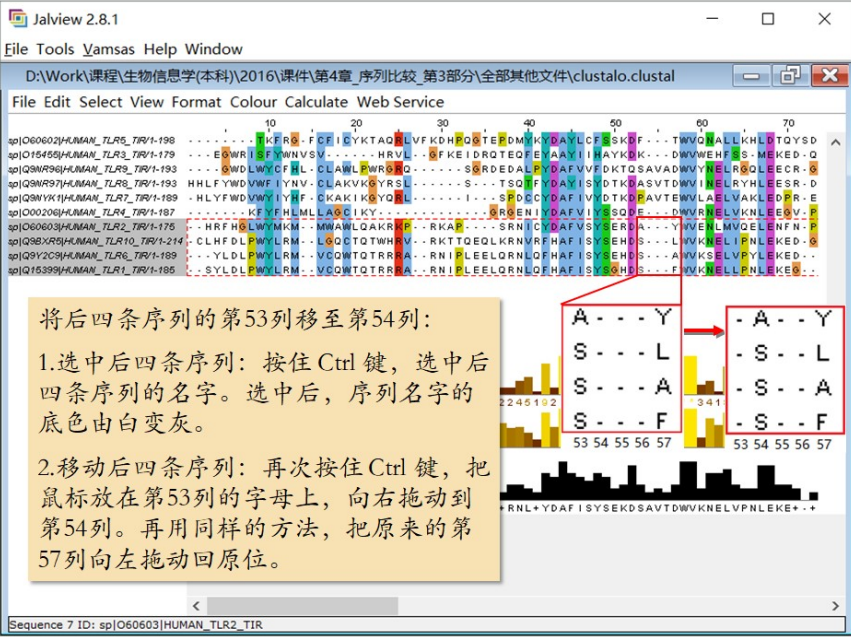
要进行如上调整,首先要同时选中要处理的 TLR2、10、6、1 这四条序列:按住 Ctrl建,用鼠标点击这四条序列的名字。选中后,序列名字的底色由白变灰,且四条序列被框入红色虚线框中。然后,再次按住 Ctrl 键,把鼠标放在任意选中序列的第 53 列的字母上,并 向右拖动至第 54 列。拖动后选中序列的第 53 列自动补充空位,原第 53 列移至第 54 列,但是从第 55 列开始往后的位置也都跟着向右移了一位。所以需要再次用同样的方法,按住 Ctrl 键,把鼠标放在任意选中序列的第 58 列的字母上,向左拖动至第 57 列的位置,以保持第53、54 列以外的位置不变。如此,便完成了手动调整。
调整外观
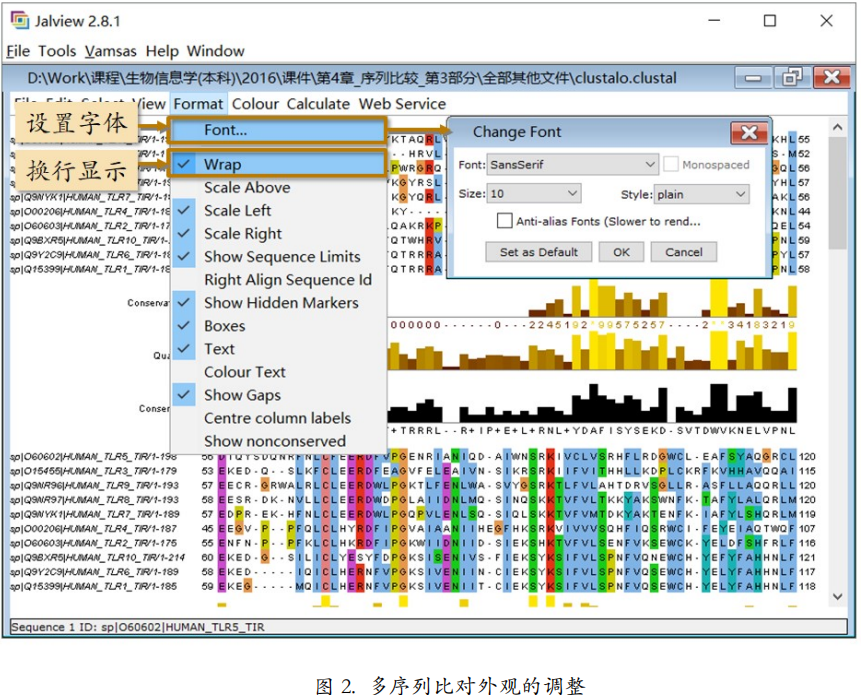
多序列比对的外观也很重要。默认情况下,多序列比对是单行显示的。由于序列长,需要拖动窗口拉条才能浏览全部。这样不利于查看分析,也不利于将导出的比对图片插入文献。 如果想要让多序列比对根据 Jalview 窗口的宽度自动换行,可以在 Format 菜单下勾选“Wrap”(图 2)。
此外,还可以通过“Font…”窗口对字体格式、大小等进行调整。
如果你只需要多序列比对,而不需要有关保守度等的注释行。可以关闭 View 标签下的 “Show annotations”选项,以达到去掉注释行的目的。
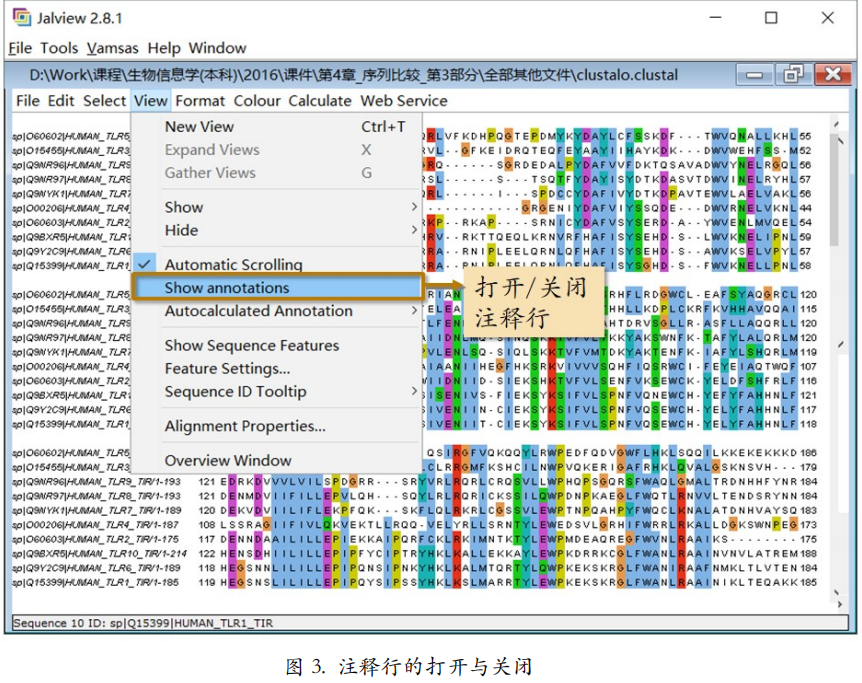
分析功能
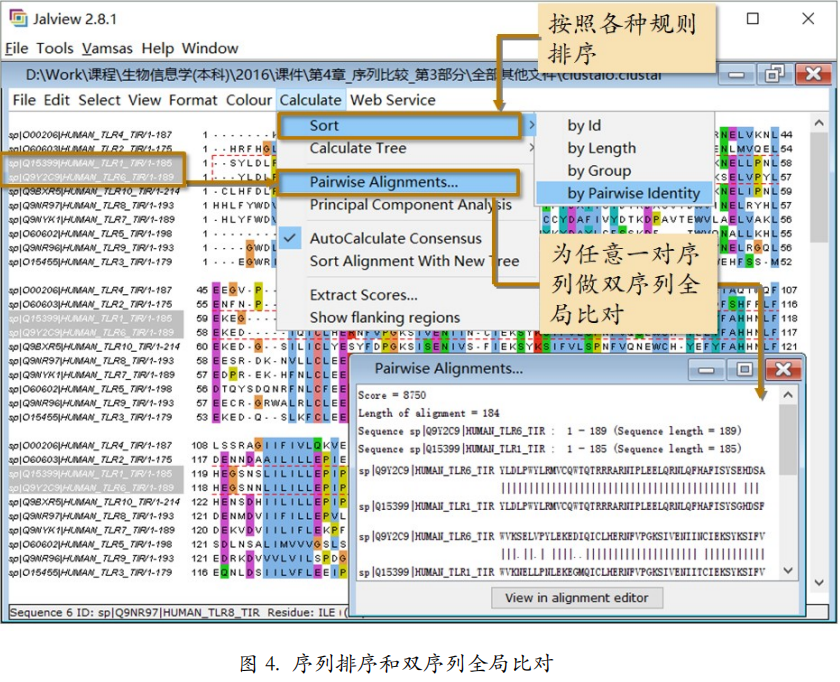
Jalview 除了有编辑多序列比对的功能还有很多分析功能。比如,
可以按照序列的名字、两两一致度或其他规则给比对中的序列重新排序以及为选中的两条序列做双序列全局比对 (图 4)
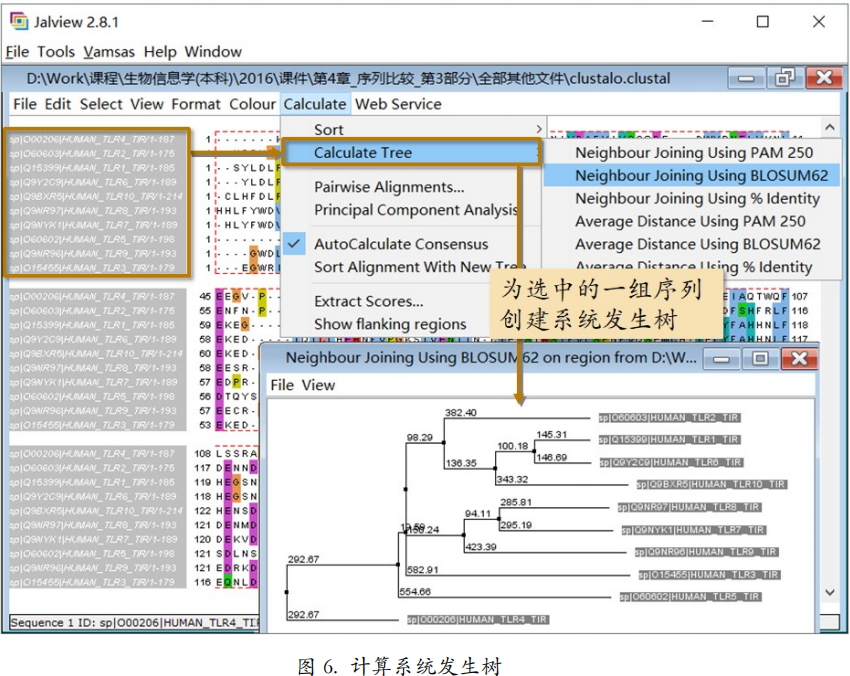
为选中的一组序列计算各种系统发生树(图 5)
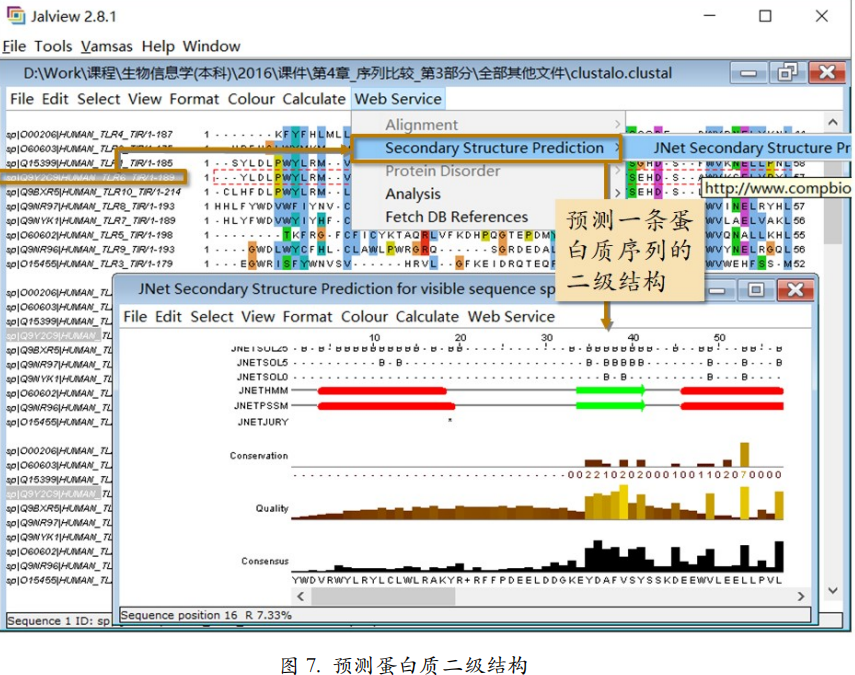
或者用在线软件为某一条序列预测二级结构(图 6)。Web service 菜单下的所有功能都需要网络支持才能运行。更多 Jalview 的功能需要大家在实践中不断摸索。
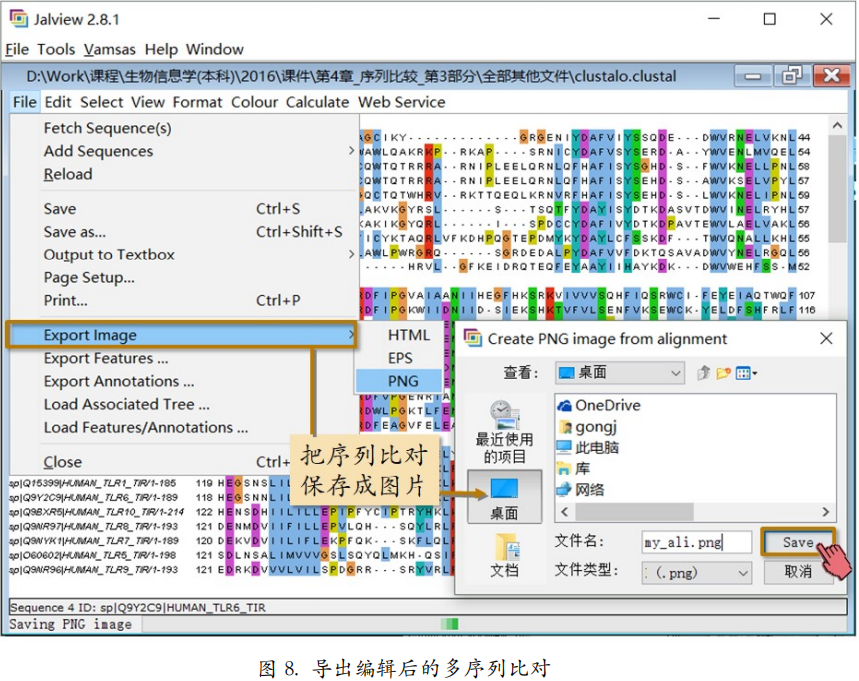
导出比对图
最后一步就是导出多序列比对为图片,插入到需要的地方:File 菜单 - Export Image -选择图片格式 - 保存(图 8)。
其他比对美化工具
除了 Jalview,还有很多比对美化工具(表 1)。Boxshade 擅长黑白制图。因很多学术期 刊只收取彩图的编辑费,所以黑白图可以节省科研经费。ESPript 的功能十分强大。MView 擅长把彩色多序列比对转换成 HTML 源代码。这样就可以将它直接插入网页,并方便以文本形式选取。
| 名称 | 网址 | 特点 |
|---|---|---|
| JalView | http://www.jalview.org | JAVA,可嵌入网页 |
| Boxshade | http://www.ch.embnet.org/software/BOX_form.html | 擅长黑白作图 |
| ESPript | http://espript.ibcp.fr/ESPript/ESPript | 功能强大 |
| MView | http://bio-mview.sourceforge.net | 擅长转换成 HTML 源码 |
多序列比对是生物数据分析中特别常用的,也是文章里经常出现的,把它做漂亮了会令你的文章增色不少。把它分析好了,就会从中挖掘出许许多多重要的信息。
3.12 寻找保守区域
序列标识图 WebLogo
保守区域
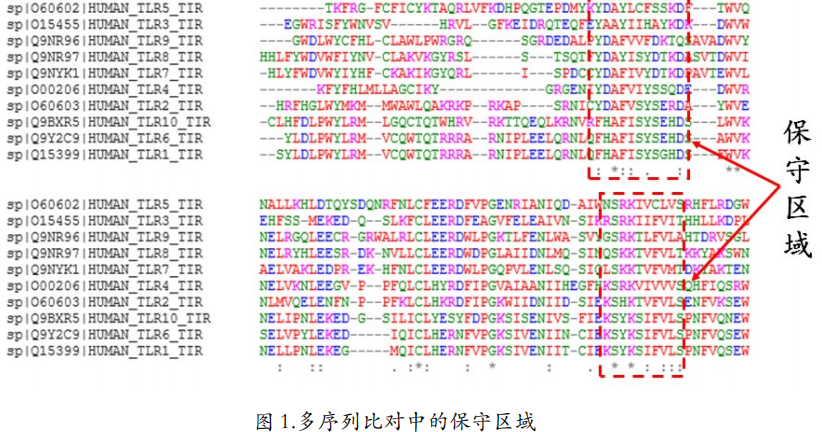
如果用一句话来描述你究竟想从多序列比对中得到什么,答案是你想要找到序列中重要的位置。说得更专业一点,就是要找到保守区域。通过多序列比对下方的星星点点可以大致发现图 1 中两个红框中的区域比较吸引眼球,因为星星点点特别多!我们稍微回忆一下这些 密码的含义:“”代表这一列残基完全相同;“:”*代表这一列残基或者相同或者相似;“.”**代表这一列残基有相似的但也有不相似的;什么都没有代表这一列残基都不相似。所以我们寻找的就是星星点点特别多的区域。当然用眼睛来数星星不那么靠谱。我们仍然需要借助软件来更好的寻来保守区域。
序列标识图
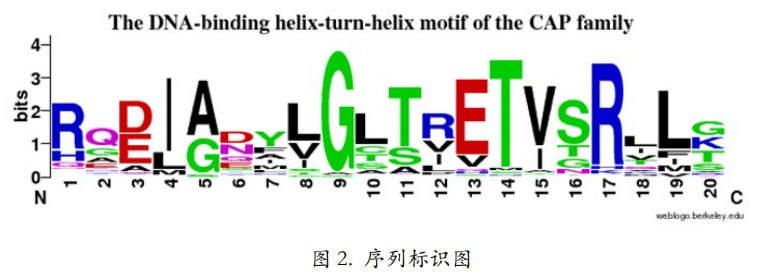
序列标识图(sequence logo)就是序列的 logo,它是以图形的方式依次绘出序列比对中各个位置上出现的残基,每个位置上残基的累积可以反应出该位置上残基的一致性。每个残基对应图形字符的大小与残基在该位置上出现的频率成正比。 但图形字符的大小并不等于频率百分比,而是经过简单统计计算后转化的结果。图2 是用一款流行的软件 WebLogo 创建的序列标识图
要创建序列标识图,首先需要一个多序列比对。多序列比对中的一列对应序列标识图中的一个位置。然后分别计算每一列中不同残基出现的频率,再根据以下公式把频率转换成高度值,最后根据高度值写出不同残基的彩色字母图形。

- 如果某一列非常保守(熵值小),字母高度就高。反之,如果某一列没有什么特征,各种残基都有出现,杂乱无章,那么就会看到一堆比较矮的字母摞在一起。
- 这里再次强调,字母的高度和 它在某一列中出现的频率成正比,但是并不等于频率。试想一下,如果字母高度就是频率的话,那么序列标识图中每个位置上字母摞起来的总高度应该是一样的,都是 100%。
- 但是从 图 2 中可以看到,序列标识图上每个位置字母摞起来的总高度是不一样的,这是因为在字母高度的计算过程中涉及了熵值。某一列中字母出现的情况越混乱(越不保守),熵值越大,字母越矮。字母出现的情况越有规律(越保守),熵值越小,字母越高。所以序列标识图可以很好的展现多序列比对中每一列的保守程度,即,它们是杂乱无章的,还有有规律可循的。并且把可循的规律图形化的展现出来。这就是我们为什么要给序列打上 logo 的原因。
WebLogo
WebLogo 是一款在线创建序列标识图的软件
主页面上点“Create your own logos”,然后输入多序列比对(图 2)。WebLogo 可以接受大多数常见的多序列比对格式。
示例文件 promoter.fasta 是一组启动子序列的多序列比对,以 FASTA 格式存储。FASTA 格式的多序列比对要求把多序列比对中的每一条序列连同插入的空位一起按 FASTA 格式书写,写完一条序列再写下一条。这和之前讲过的 Clustal 格式不太一样。
在序列输入框的下方可以设置不同参数,以定义序列标识图的样式,比如设置序列标识图的创建范围、定义字母的颜色方案等。保持所有参数默认,点“Create WebLogo”。
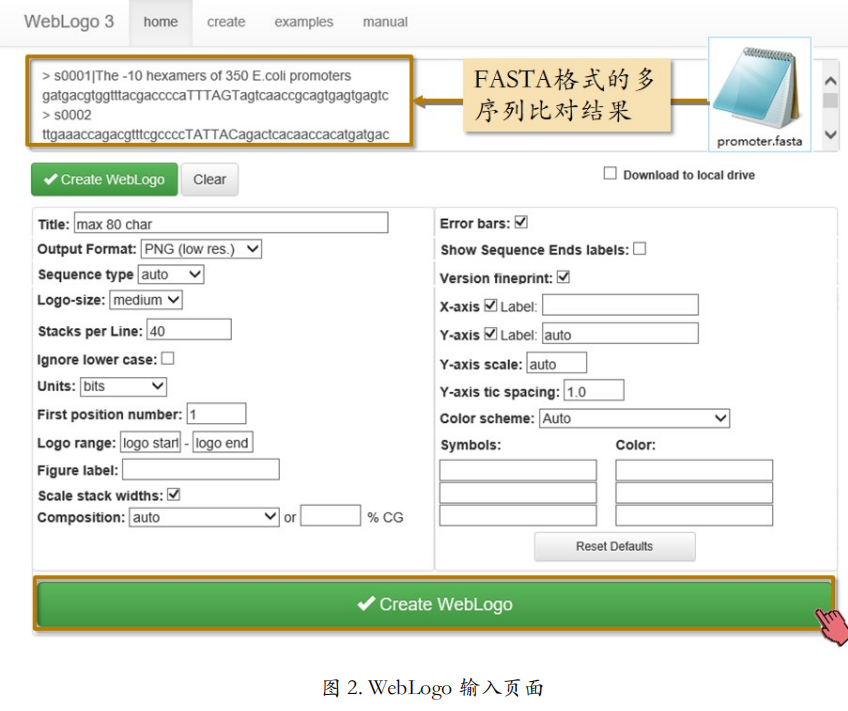
图 3 为创建出的序列标识图。从图中可以清晰的看到:输入的这些启动子序列上 TATA-Box 的共有特征序列,以及它们出现的位置
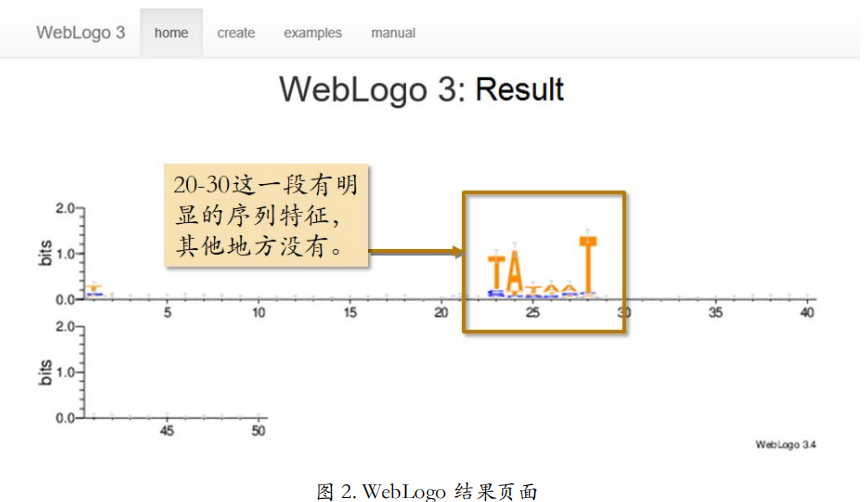
序列基序 MEME
序列基序
在核酸或蛋白质序列中存在一些有特定模式的序列片段,这些片段称为序列的基序(motif)。
序列的基序与生物功能密切相关。比如,发生 N 糖基化位点的基序:发生糖基化的天冬酰胺后面一定紧跟一个脯氨酸以外的氨基酸,再紧跟丝氨酸或者苏氨酸,再紧跟一个脯氨酸以外的氨基酸。
这个特定模式可通过正则表达式来规范描述,也可以通过序列标识图来直观描述。基序的发现要通过大量相关序列的分析。MEME 就是一款可以自动从一组相关的核酸或蛋白质序列中发现序列基序的软件。
MEME
MEME 是 The MEME Suite 在线软件套装中的一员。MEME 的使用非常简单,只需要将待分析的序列上传即可(图 1)。而且,上传的序列为原始序列, 不需要提前为它们做多序列比对。你也可以指定返回排名前几的基序。MEME 的等待时间 稍长,大约 10 分钟以上,所以最好留下邮箱
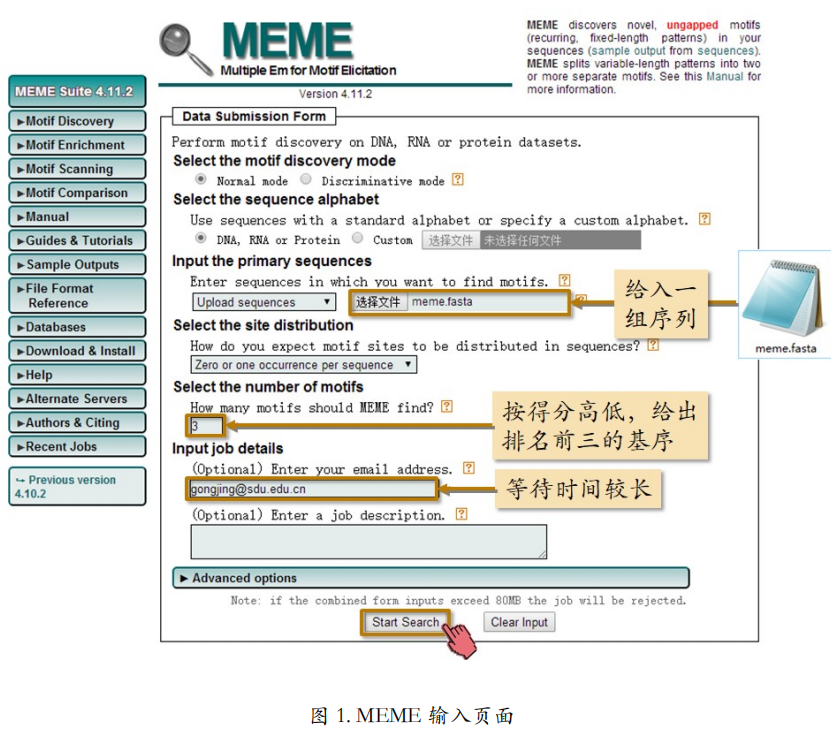
MEME 的返回结果被保存成各种格式:HTML、XML、test 等。便于在线查看的是“MEME HTML output”,即网页格式。
网页格式的 MEME 结果页面中,给出了找到的排名前三的基序(图 2)。它们以序列标识图的形式展现出来。同时还提供这三个基序在每条序列中的大体位置。

如果要进一步了解某个基序,可以点击序列标识图右侧的“More”下面的“↓”箭头,以查看详细(图 3)。点击后,会得到大比例序列标识图,以及该基序在每条序列中对应的序列片段和它们出现的具体位置。

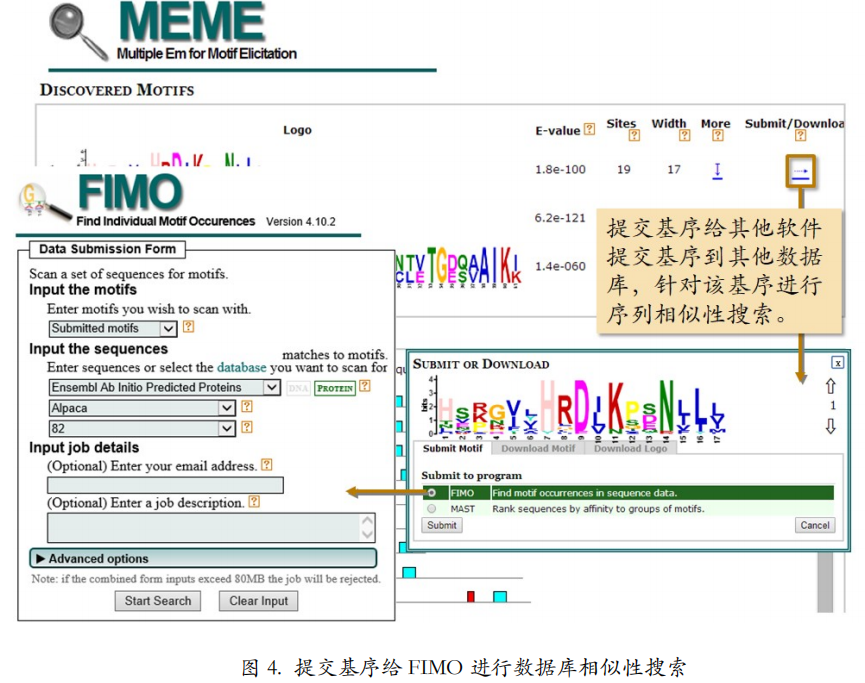
此外,还可以点击序列标识图右侧的“Submit/Download”下面的“→”箭头(图4),将某个基序提交至各种数据库,并进行针对该基序的序列相似性搜索,已找到数据库中含有该基序的序列,进而推测该基序的功能。这步操作是通过 The MEME Suite 软件套装下的另一个软件 FIMO 来实现的。
寻找保守区域:PRINTS指纹图谱数据库
蛋白质指纹
蛋白质的指纹是指一组保守的序列基序,用于刻画蛋白质家族的特征。这些基序由多序列比对结果获得,且它们在氨基酸序列水平上是不相邻的,但是在三维结构中可能紧密地结合在一起。
目前,科学家已经对现有的蛋白质序列进行了充分的研究,而且早已发现并总结了这序列上的重要基序。相关成果汇入了PRINTS蛋白质序列指纹图谱数据库(http://www.bioinf.manchester.ac.uk/dbbrowser/PRINTS/)。
PRINTS数据库存储了目前已发现的绝大多数蛋白质家族的指纹图谱。对于一个陌生的蛋白质,只要看看它的序列是否符合某个蛋白质家族的图谱就可以对它进行分类并预测它的功能
PRINTS数据库
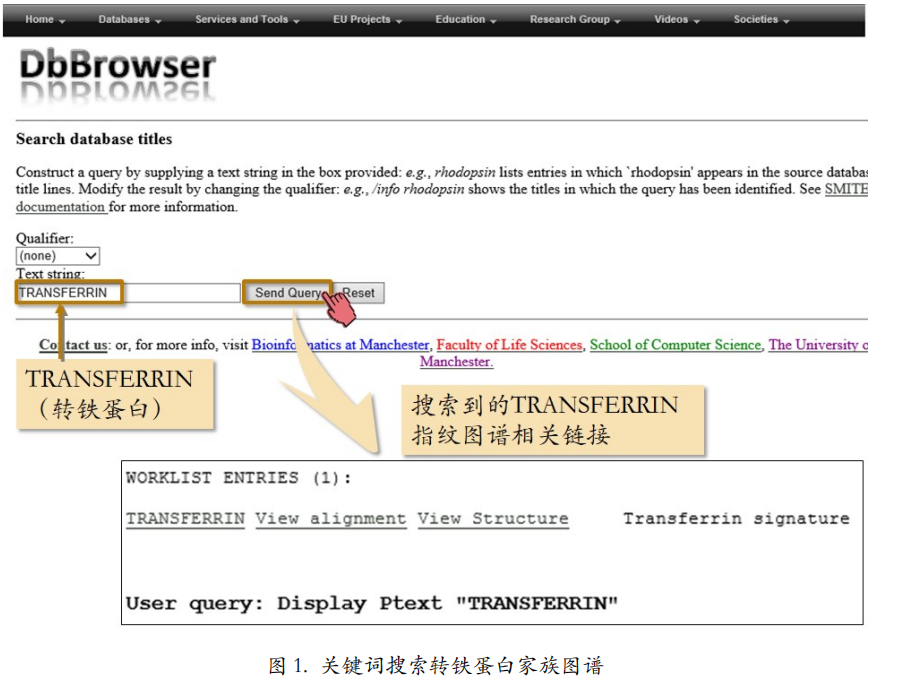
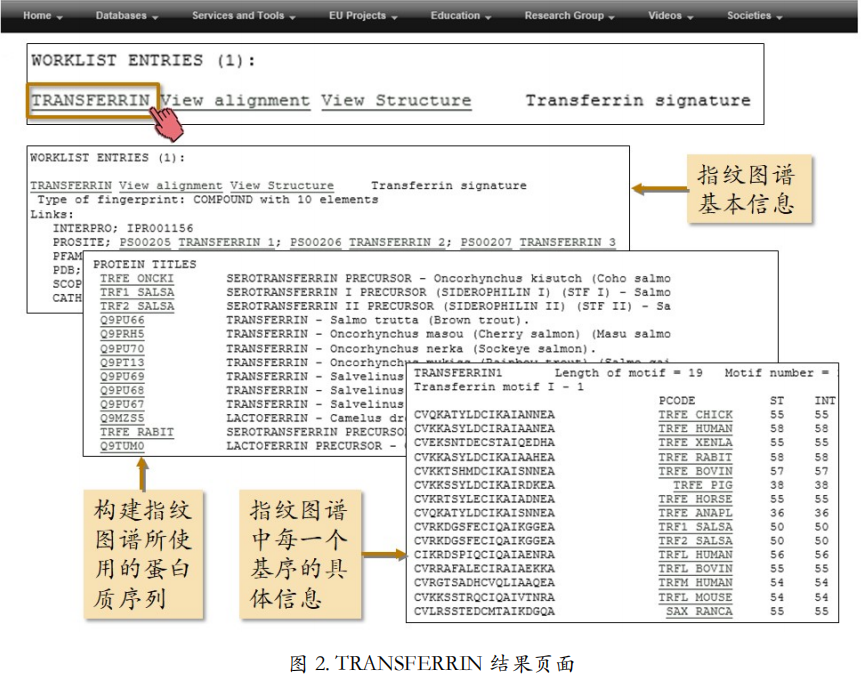
- 要浏览PRINTS数据库,可以输入数据库编号、关键词、或标题等以查找某一个指纹谱。比如点击“By text”通过关键词搜索(图1)。输入条中输入“TRANSFERRIN”,也是搜索转铁蛋白家族的图谱。搜索返回转铁蛋白家族的指纹图谱链接。
- 点击结果页面中的“TRANSFERRIN”链接后,会显示包括指纹图谱的基本信息、与其他数据库之间的交叉链接、构建指纹图谱所使用的蛋白质序列、以及指纹图谱中每个基序等具体信息(图 2)。
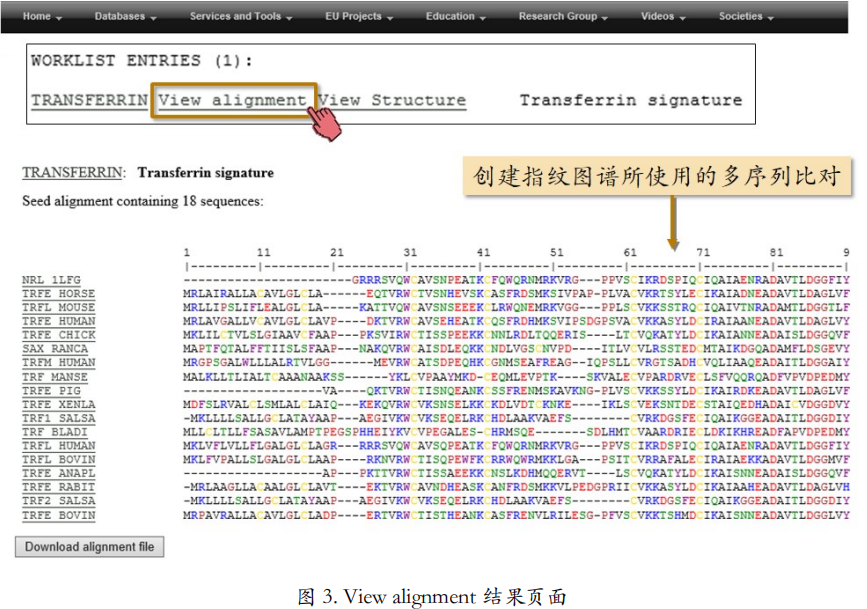
- 点击“View alignment”链接后,可以看到创建指纹图谱所使用的多序列比对(图 3)。
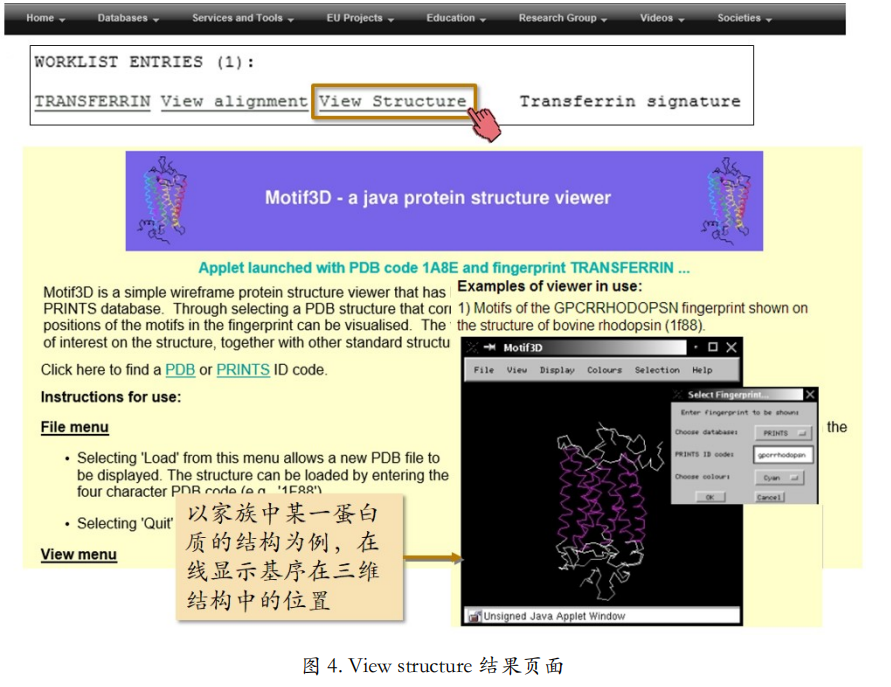
- 点击“View structure”链接后,网页会打开一个三维视图插件,并以该家族中某一特征蛋白质具有的三维结构为例,在线显示指纹图谱中各个基序在三维结构中的位置(图 4)。从该三维结构图中可以看出,紫色的基序在氨基酸序列水平上并不相邻,但是在三维空间结构中是紧密联系在一起的,并形成蛋白质的重要功能区。
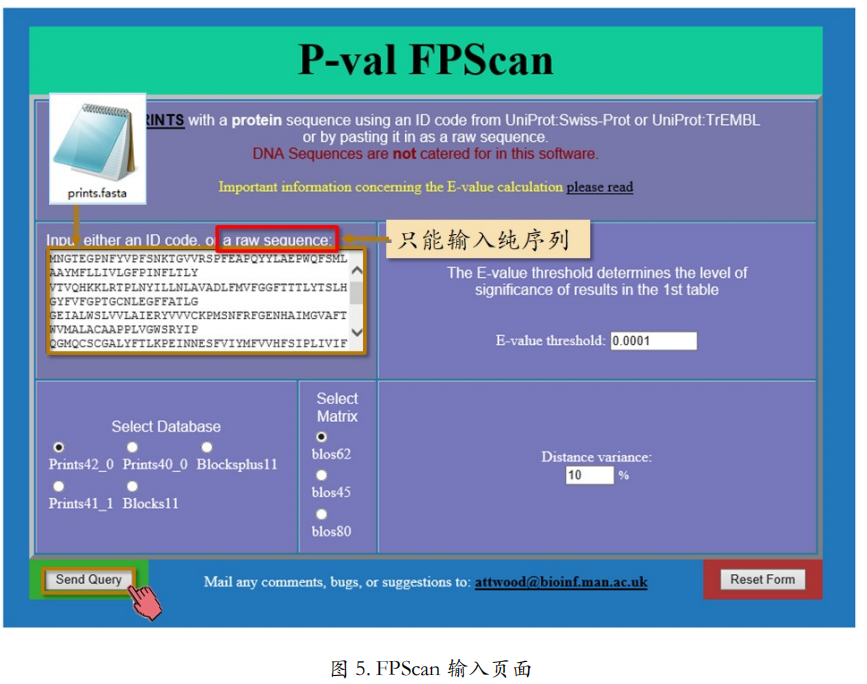
除了浏览某一指纹图谱,PRINTS 还提供指纹匹配服务。也就是搜索某一序列所匹配的指纹图谱。此功能通过 PRINTS 主页也上的“FPScan”链接实现(图 5)。注意输入的待搜索序列只能是“a raw sequence”,也就是纯序列。换言之,FASTA 格式中带大于号的第一行不能拷贝进输入框。示例文件 prints.fasta 请从课程附件中下载。
提交后返回的结果页面中,跟输入序列匹配的指纹图谱,根据匹配得分的高低被排列出来(只列出前十名)(图 6)。此外,还单独列出了排名前三的指纹图谱。由此可知,得分最高的是视紫红质家族的指纹图谱。
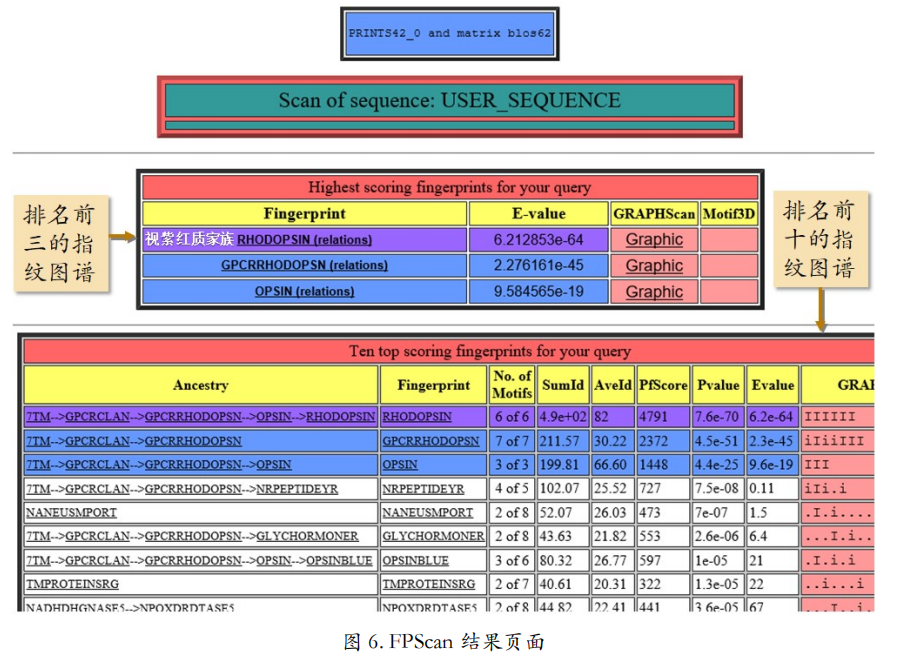
点击排名第一的视紫红质家族的“Graphic”链接,可以得到该家族指纹图谱中各个基序在输入序列中所匹配的位置(图 7)。结果页面的下部还提供了视紫红质家族的 6 个基序在输入序列中所对应的具体序列片段。由此,可以推测,输入序列属于视紫红质家族,并具有该家族蛋白质的主要功能。事实上,输入序列确实是从 UniprotKB 数据库中下载的一条羊的视紫红质的序列(P02700)。
第四章 分子进化与系统发生
4.1 进化的故事
拉马克与用进废退
拉马提出进废退理论。他说生物经常使用的器官会逐渐发达,不经常使用的器官会逐渐退化。而且这种后天获得的性状是可以遗传的,因此生物可以把后天锻炼的成果遗传给下一代。拉马克举了长颈鹿的例子。他说,长颈鹿原本脖子没那么长,但是为了吃到高处树叶不断的伸长脖子,久而久之,他的脖子越伸越长,并且把长脖子这个后天获得的性状,遗传给了下一代。再有鼹鼠一年到头的在地下打洞。因为地下漆黑一片,打洞只要鼻子闻,根本用不着眼睛看,久而久之,都成了瞎子。再有鸟吃饭都不嚼直接咽,所以鸟嘴里都没牙,而哺乳动物比如猫要撕咬食物,所以尖牙利齿。
拉马克主义提出后,生物界支持声和反对声此起彼伏。先来听听反对他的声音。德国科学家魏斯曼做了一个实验。他把老鼠的尾巴都切断,之后再让没有尾巴的老鼠互相交配,生 出的下一代老鼠依然是有尾巴的。然后再把子代老鼠的尾巴切断之后交配,生出的下一代老 鼠依然是有尾巴的。实验一直重复至第 21 代,但是老鼠的尾巴依然长长的,一点儿都没短。于是魏斯曼说拉马克是错的!
达尔文与自然选择
拉马克主义争论了几十年之后,达尔文时代终于来临了。或许你要问的第一个问题应该是“达尔文究竟做了什么?”。除非你与世隔绝,否则你一定听说过查尔斯-达尔文。但在研究出革命性的进化论之前,达尔文的生活却与生物学没什么关系。最初,他从医学院辍学,因为他总是晕血。之后他又差点成为一位神职人员。鉴于他有望成为神的仆人,那么达尔文 不是提出进化概念第一人的事实就不那么令人惊讶了。在大学里,达尔文对自然注意十分着迷。但是要以此作为事业的话,他需要习惯海上的颠簸。1835 年他抵达了加拉帕戈斯群岛, 在那里,自然的独特本质撼动了达尔文的世界。一切始于加拉帕戈斯地雀,在这一列岛中,每个岛上的地雀都长着独特的喙部。独特的喙部似乎都是为了适应不同的食物而生,不管是种子、仙人掌、还是幼虫。达尔文认为这绝不可能是巧合,于是开始调查研究生物的适应性。这项研究话费了 20 年才得以完成。然而他得出的结论却与当时盛行的神创论中关于生物起源的一切观点相悖。
达尔文认为,所有的生物物种都存在趋利的适应性变化。并且这些适应性变化会通过一种他称之为“自然选择”的过程遗传下去。而大自然是数百万年间推动演变 进化的唯一力量。这个理念让达尔文成为了现代生物学之父。对于一个渴望成为牧师的人来说,这个称谓也不赖。进化无时无刻不在发生,这一理念产生后,人们就想找到一种机制。 而这就是达尔文的贡献所在,也是他为何会花费如此长的时间进行思考、收集证据以及整理 入书。达尔文认为“物竞天择”意味着,存在一种最初的生物,之后通过某种方式得到了改 良。如果环境对你施加压力,压力有可能是捕食者的威胁或类似的情况,那些通过某种手段 生存下来,并且繁衍后代的生物,他们的后代也能生存下来,并且继续繁衍生息。因此,如 今我们所见到的动植物都拥有很强的适应性。他们所表现出来的适应性使得他们成功存活,并继续繁衍下一代。
4.2 基本概念
如何研究进化
传统方法
化石
这是进化最直接,最确凿的证据。假如我们可以获得某个物种自诞生之日起每隔一百年的所有完整化石, 那么我们就可以看出这个物种是怎么一点一点演变来的。但是非常可惜的是,生物化石很零散,不完整。人类得到生物化石是个极其偶然的事件。
利用比较形态学、比较解剖学和生理学等手段,确定大致的进化框架
这种方法比第一种方法更容易实现。但是这种方法仅局限于大致 的框架,很多细节是存在争议的。只能是仁者见仁智者见智,因为用肉眼观察存在误差。还有一个问题就是伦理学问题。
分子进化方法
- 利用软件,从分子水平上构建物种的进化树。这里说的分子水平是指 DNA、RNA、以及蛋白质序列。通过前几章的学习,我们知道随着测序技术的发展,这种分子水平上的信息量大而丰富,获取十分便捷。当然有优点自然就有缺点。缺点就是准确度依赖软件的优劣及参数的设置。随着相关软件的不断升级,这个缺点也再逐渐被克服。
- 分子进化理论:
- 分子进化理论是 1962 年美国科学家 Linus Pauling提出的。
- 这种理论与传统研究方法的最大区别是,它研究的是 DNA、RNA 以及蛋白质序列这些分子水平上的信息,而不是物种的外在特征。并且基于某一个特定的分子在不同物种中的序列差异来构建系统发生树。
- 此外,分子进化有两个基本的假设条件,只有接受这两个假设,分子进化理论才能得以实施。
- 第一、DNA、RNA 或蛋白质序列包含了物种的所有进化史信息。
- 第二是分子钟理论。这个理论说的是,一个特定基因或蛋白质的进化变异速度在不同物种中是基本恒定的。所谓变异速度是指一定时间内不同碱基或氨基酸突变的个数。这 进化变异速度被认为是恒定的,跟物种没有关系。所以,拿蛋白质来说,两个蛋白质在序列上越相似,他们距离共同祖先就越近。
- 分子钟理论是进化研究领域被普遍认可的理论,但是至今也没有直接的证据证实。
不同的同源
同源Homologs
- 来源于共同祖先的相似序 列为同源序列。
- 也就是说,相似序列有两种,一种是来源于共同祖先的,那么他们可以叫同源,另一种不是来源于共同祖先的,那么他们尽管相似也不能叫同源。第二种情况出现的概率虽然低,但还是存在的,所以相似序列并不一定是同源序列。
- 同源又分为三种,直系同源,旁系同源和异同源。
直系同源(Orthologs)
来自于不同物种的、由垂直家系,也就是物种形成,进化而来的基因,并且典型的保留与原始基因相同的功能。也就是说,随着进化分支,一个基因进入了不同的物种,并保留了原有功能。这时,不同物种中的这个基因就属于直系同源。
旁系同源(Paralogs)
是指在同一物种中的来源于基因复制的基因,可能会进化出新的但与原功能相关的功能来。
基因复制产生了两个重复的基因,多出来的这个有几种命运,一 个是又丢了。复制出来发现没有用,又删了。另一种命运是演化出了新的功能。如果这个新功能是往好的方向发展,就会被保留下了,如果是往不好的方面发展,就会被自然选择淘汰。 还有一种命运,就是被放置不用。复制出来以后,又加了个终止子,既不表达,也不删除, 搁那里搁着不管,成了伪基因。被保留下来的具有新功能的基因与另一个复制出来的基因之间就是旁系同源。
异同源(Xenologs)
通过水平基因转移,来源于共生或病毒侵染所产生的相似基因。异同源的产生不是垂直进化而来的,也不是平行复制产生的,而是由于原核生物与真核生物的接触,比如病毒感染,在跨度巨大的物种间跳跃转移产生的
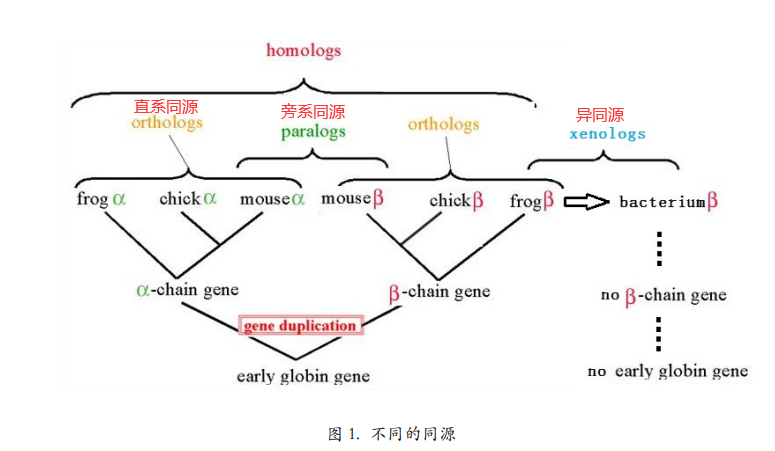
不同的同源,概念很容易混淆。图 1 清楚的描述了各种同源之间的关系。首先,有个早期的球蛋白基因,它通过基因复制,形成了α球蛋白基因和β球蛋白基因。后来随着进化,这两种复制产生的基因也存在于不同的物种中。
其中某一物种里的,比如老鼠里的α球蛋白基因和β球蛋白基因就属于旁系同源。
而某一个基因在不同物种中,比如青蛙里的α球蛋白基因和鸡里的α球蛋白基因就属于直系同源。
再比如,某个细菌,它没有早期的球蛋白基因,也自然没有β球蛋白基因,但是通过与青蛙的共生,发生了基因水平转移。于是它从某一天就起有了β球蛋白基因。那么这个细菌的β球蛋白基因和青蛙的β球蛋白基因就属于异同源。
特别注意!!!
同源只是对性质的一种判定,只能定性描述,不能定量描述。所以“同源性等于 80%”这种 说法是错误的!
“树状”还是“网状”
越来越多的细菌和动植物的基因组测序显示,基因并不是简单遗传给生命树上的个别枝条,它们还在物种之间以不同的进化路径转换,其结果是一个杂乱无章的“生命网”。这里 要区分“树”和“网”的概念。在计算机科学领域,树的定义规定,树上从一个点到另一个点的路径只有唯一的一条。而当两点之间的路径个数≥2 的时候,就形成了网。编织生命网 的要素之一就是水平基因转移。水平基因转移,是指生物将遗传物质传递给其他细胞而非其子代细胞的过程。
早在 1993 年,就有生物学家提出细菌的基因排序不是树状的,而是网状的。1999 年,美国《科学》杂志发表言论说:“生命进化树并不是真实存在于自然界中的,而是人类用来规划自然界的一个理论。”但是,有研究者运用更多的研究捍卫达尔文的观点,认为所谓网状的进化论是理想化、不切实际的想法。
“树状”和“网状”的辩论在 2006 年正式拉开帷幕。位于德国海德堡的欧洲分子生物实验室(EMBL)派出由皮尔·博克领导的工作组,研究了来自细菌、古细菌以及真核细胞 的 191 个基因组。他们发现,其中 31 组基因没有任何迹象表明曾经被水平转移过,和尚未 完善的“树”相近。但是,来自德国杜塞尔多夫大学的达冈和马汀教授认为,31 组的结果 不能够证明什么,这个数字太小。 2008 年,达冈和他的团队研究了 181 个基因组,发现 80% 的基因组存在水平基因转移,即网状树。
此外,杂交也可能是物种进化的有力驱动。来自伦敦大学的生物进化学家詹姆斯·马里特说:“杂交是非常普遍的现象,有 1/10 的动物都是杂交的。”2008 年,美国得克萨斯大学 的科学家在包括家鼠、野鼠和非洲爪蛙在内的 8 种动物的基因组中发现了一种奇特的 DNA。 这是鸡、大象和人类所没有的 DNA,这说明它是一些动物通过异种交配形成的。几年前,科学家也曾在牛体内发现蛇的 DNA。此外,鱼类、昆虫和植物中也都曾发现水平基因转移 现象。这些新发现意味着,有一些物种的进化是呈现树状的,而有一些却不是,所以用达尔文的进化枝条来连接物种似乎过于简单了。
4.3 系统发生树
系统发生树的样子
研究分子进化所要构建的系统发生树(Phylogenetic tree),也叫分子树。
作用
首先来看从系统发生树上我们都能研究出什么?
对于一个未知的基因或蛋白质序列,可以利用系统发生树确定与其亲缘关系最近的物种。比如你得到了一个新发现的细菌的核糖体 RNA,你可以把它跟所有已知的核糖体 RNA 放在一起,然后用他们构建一棵系统发生树。这样就可以从树上推测出谁和这个新细菌的关系最近。
系统发生树还可以预测一个新发现的基因或蛋白质的功能。以基因为例,如果在树上与新基因关系十分密切的基因的功能已知,那么这个已知的功能可以被延伸到这个新基因上。
构建系统发生树还有助于预测一个分子功能的走势。也就是从树上可以看出某个基因是正在走向辉煌还是在逐渐衰落。
最后,系统发生树还能帮助我 们追溯一个基因的起源。甚至当它从一个物种“跳”到另一个物种上,也就是发生了水平基因转移时,系统发生树都可以很好的展示出来
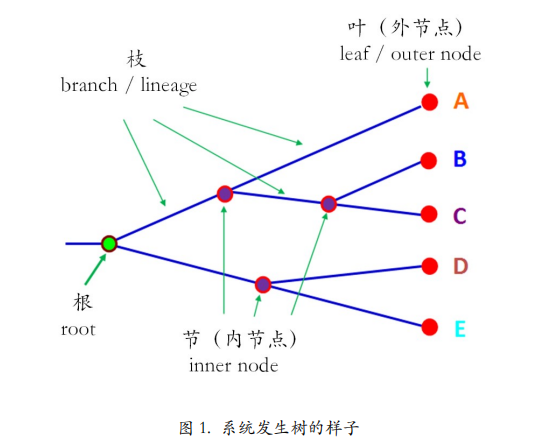
介绍
树是从根(root)长出来的。从根延伸出的树枝就叫枝(branch/lineage)。枝上有分叉,分叉的地方就叫节(node)。枝的顶端顶着的就是叶(leaf)。根、节和叶都可以叫做节点(node)。但是叶后面不再有枝了,是最外面的节点,所以叫外节点(outer node)。而节的前后都有枝,所以叫内节点(inner node)。
根是一切的起源,习惯上就叫根。根和节都表示理论上曾经存在的祖先,叶子是现存的物种。这一点很重要!比如我们要研究某个基因,于是搜集了很多物种的这个基因的序列,用它们构建了一棵系统发生树。搜集到的物种都出现在叶子上,也就是外节点上,没有在内节点上的。内节点上都是理论上曾经存在过的共同祖先,现在已经不存在了!此外,枝子的长短也是有意义的,我们后面再讲。
用软件创建出的系统发生树有各种形状。圆形的,方形的,三角形的等(图 2)。从生物学意义上没有任何区别,只是表现形式不同而已。根据建树所用序列的多少来选择不同形状的树。如果序列非常多,那么圆形的看上去就要比方的或者三角的舒服得多,便于在文献里排版。系统发生树上从任何一个点发出的枝子围着这个点旋转都不改变树的生物学意义, 只是视觉上有点儿差别而已。所以,旋转之后的两棵树是等价的,生物学意义完全相同。
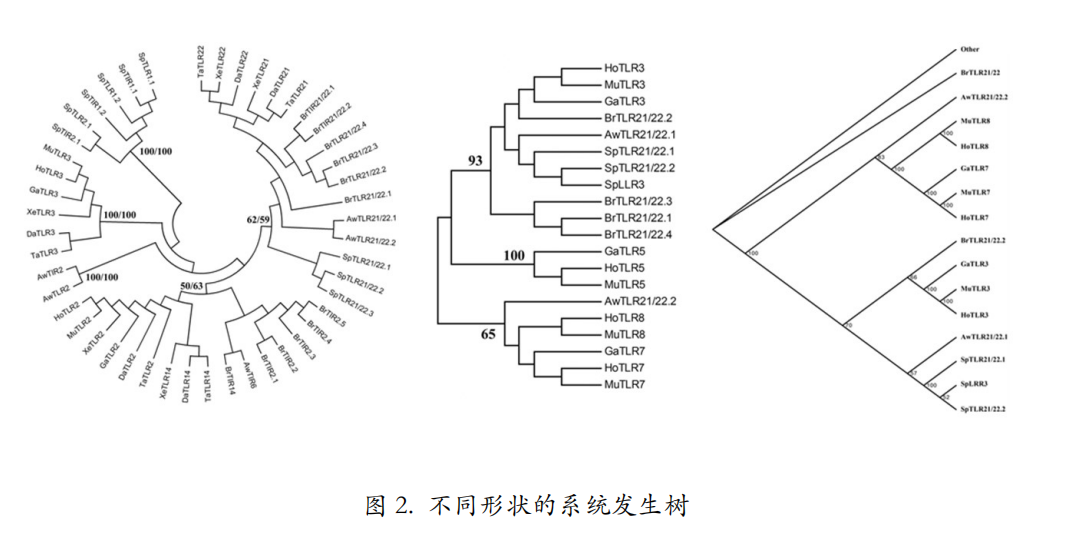
系统发生树的种类
有根树与无根树
系统发生树还分为有根树和无根树(图 1)。顾名思义,有根树就是有根,无根树就是无根。
其实两者是可以互换的。如果我们按住无根树上某一个点,然后用把梳子将树上所有 的枝条都以这个点为中心向右梳理,就能把它梳成有根树的样子。按住的这个点就是根。所 以对于一棵树来说,根的位置是主观的,你想让他在哪它就在哪里。但是你不能随意指定哪个内节点当根,毕竟根有其自身的生物学意义,它应该是所有叶子的共同祖先。那么我们如何确定根的位置呢?可以通过外类群(outgroup)来确定,从而把无根树变成有根树。
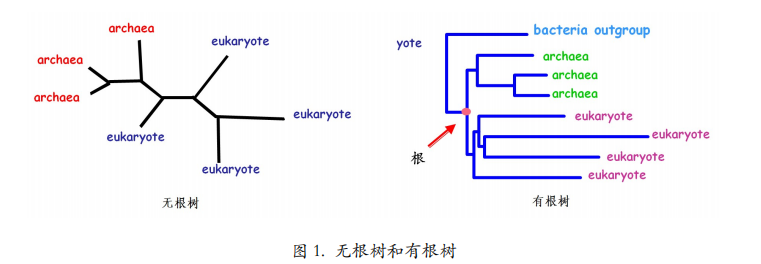
有根树反映了树上基因或蛋白质进化的时间顺序,过分析有根树的树枝的长度,可以了解不同的基因或蛋白质以什么方式和速率进化。
而无根树只反映分类单元之间的距离,而不涉及谁是谁的祖先问题。
做有根树需要指定外类群。所谓外类群,就是你所研究的内容之外的一个群。比如你要分析某一个基因在不同人种间的进化关系,那就可以额外选择黑猩猩加入进来,作为外类群一同参与建树。或者你要分析哺乳动物,那就可以选鳄鱼、乌龟之类的。总之,保证外类群在你要研究的内容之外,但又不能太远。外类群可以不只是一个物种,而是多个,但也不要太多,两三个即可。为什么有了外类群之后,做出来的树就是有根树了呢?因为你知道外类群和你研究的内容一定不是一伙的,所以外类群分支出的那个内节点就是根。
物种树
1998 年,伍斯提出了一个涵盖整个生命界的系统树(图 2),也叫物种树。之后这棵树被后人不管的补充,不断的修改,不断的完善,变得无比常庞大。物种树勾画了生物进化的 大致轮廓。从完全版的物种树上,可以找到目前人类已发现的所有有生命的东西。
我们这里讲的分子树跟物种树是有本质区别的。物种树是基于每个物种整体的进化关系,也就是基于整个基因组构建的,而分子树是基于不同物种里某一个基因或蛋白质序列之间的关系构建的。那么一个分子树表达出来的各物种之间的关系就可能与物种树完全不同。此时,说明这个基因经历了特殊的进化故事。也许是受到了特殊环境变化的影响,也许是发生了水平基因转移等等。总之,这种区别的出现是很有研究价值的。
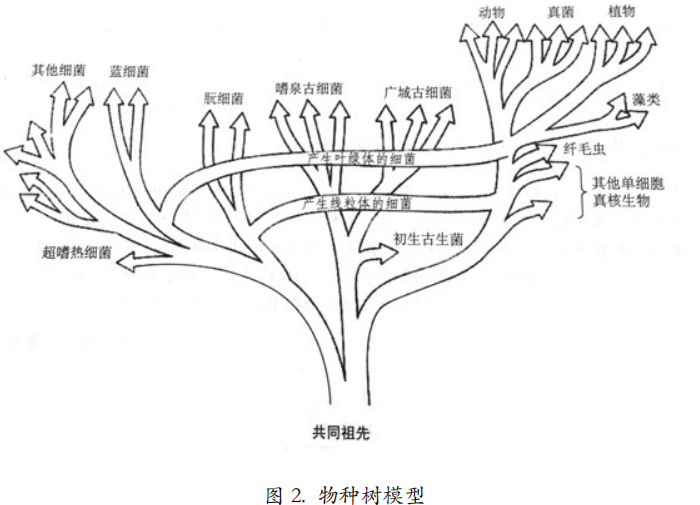
4.4 系统发生树的构建
构建方法
括非加权分组平均法 (Unweighted Pair Group Method with Arithmetic mean,UPGMA),
最近邻居法(Neighbor Joining method,NJ)
最小二乘法(Generalized Least Squares,GLS)
最大简约法 (Maximum Parsimony,MP)
最大似然法(Maximum Likelihood,ML)
贝叶斯推断法(Bayesian Inference,BI)
计算速度:基于距离的方法(UPGMA, NJ)> 最大简约法 > 最大似然法 > 贝叶斯法
计算准确度:贝叶斯法 > 最大似然法 > 距离法 > 最大简约法 > 基于距离的方法(UPGMA, NJ)
最大似然法最实用,5因为这种方法无论从速度还是准确度都比较适中。
最近邻居法虽然算得快,但是当序列多,彼此差别小的时候,这种方法不适合。
最大简约法,似乎是个掉空里的方法,高不成低不就,所以很少有人使用。
贝叶斯法不是所有的建树软件都提供,算法开发上还有待提高,而且计算时间过长。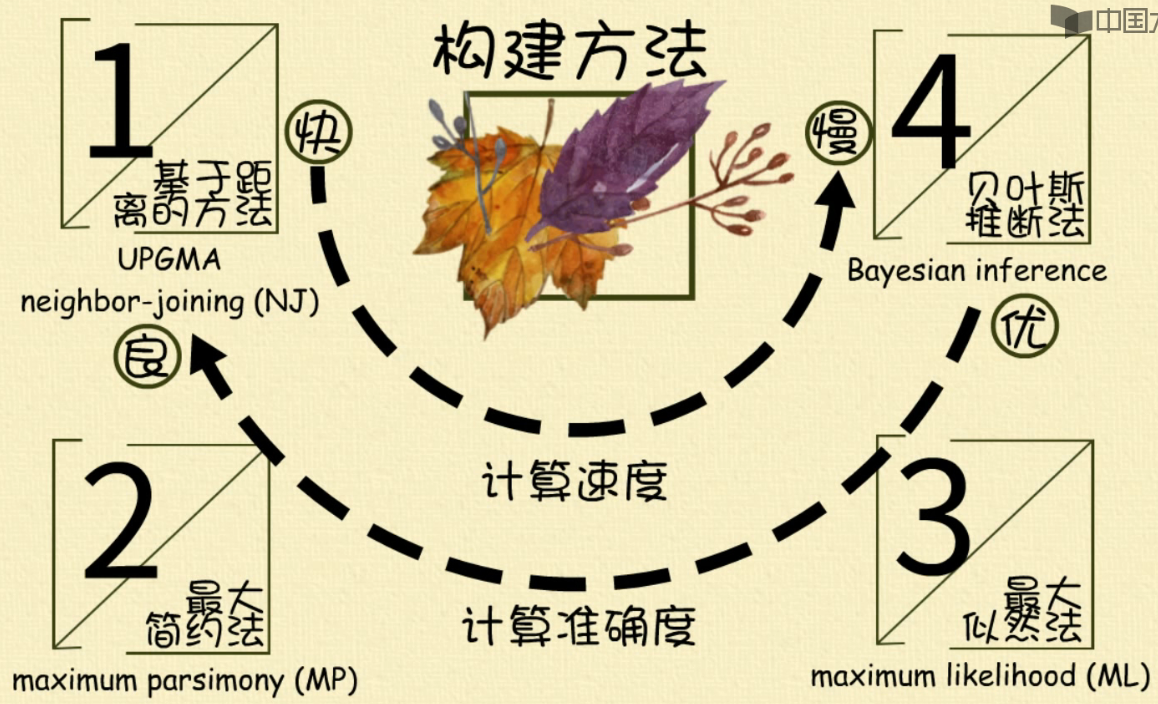
建树软件
| 软件 | 说明 | 网址 |
|---|---|---|
| PHYLIP | 免费的、集成的进化分析工具 | http://evolution.genetics.washington.edu/phylip.html |
| MEGA | 图形化、集成的进化分析工具 | http://www.megasoftware.net/ |
| PAUP | 商业软件,集成的进化分析工具 | http://paup.csit.fsu.edu/ |
| PHYML | 最快的 ML 建树工具 | http://www.atgc-montpellier.fr/phyml/ |
| MrBayes | 基于贝叶斯方法的建树工具 | http://mrbayes.csit.fsu.edu/ |
如果构建的系统发生树要用于发表生物信息学领域的文章,需要两种以上的构建方法锁定同一个结果才能审稿通过。如果是用于发表以生物实验为主的文章用一种构建方法就可以了。
UPGMA法
虽然软件可以快速自动地完成系统发生树的构建,但是对于基本算法的了解还是必不可少的。以非加权分组平均法(UPGMA 法)为例,介绍如何通过计算所有序列两两间的距离, 再根据距离远近构建系统发生树。序列两两间的距离可以用双序列比对得出的一致度/相似度代表,或用其他简化值代替。
比如,有如下 A、B、C、D 四条序列:

在接下来的例子里,我们用序列间不同的碱基数目作为序列间遗传距离的度量。首先,计算出每两条序列间有几个碱基不同,并以用矩阵的形式记录下这些距离。
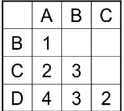
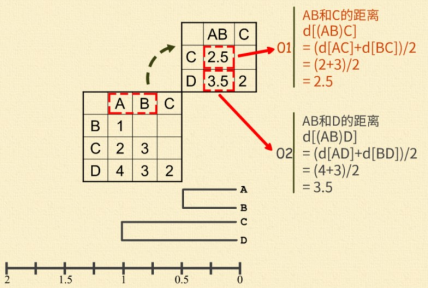
找出距离最小的一对序列。A 和 B 之间的距离最小,d[AB]=1。然后将 A 与 B 合并聚集,其分支点为 d[AB]/2=1/2=0.5。即,A、B之间的距离等于 1,从中间折叠后每边各 0.5。
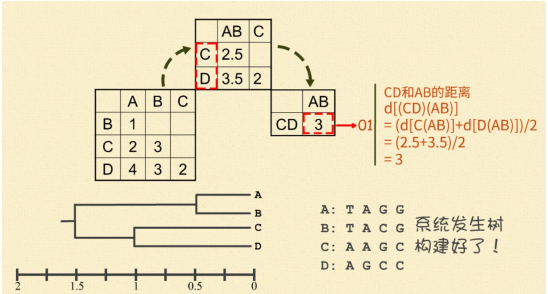
现在,把(AB)看成一个整体, 分别计算它们与 C 和 D 的距离。(AB)和 C 的距离等于 A 和 C 的距离加上 B 和 C 的距离除以 2,即,d[(AB)C]=(d[AC]+d[BC])/2 =(2+3)/2=2.5。同样,(AB)和 D的距离等于 A 和 D 的距离加上 B 和 D 的 距 离 除 以 2 , 即 , d[(AB)D]=(d[AD]+d[BD])/2=(4+3)/2=3.5。据此,计算出新的距离矩阵,并找出新矩阵中最小的距离。C 和 D 之间的距离最小,d[CD]=2。将 C 和 D 进行合并聚集,其分支点为 d[CD]/2=2/2=1。
接下来,把(CD)看成一个整体,计算它们与(AB)之间的距离。(CD)与(AB)之间的距离等 于 C 和 (AB) 的 距 离加上D 和 (AB) 的 距 离 除 以 2 , 即 , d[(CD)(AB)] = (d[C(AB)]+d[D(AB)])/2 =(2.5+3.5)/2=3。最后,将(AB)与(CD)进行合并聚集,归为一类,分支点为 d[(CD)(AB)]/2=3/2=1.5。这样,A、B、C、D 四条序列的系统发生树就构建好了。树上,枝的长短直接反应了它们与共同祖先的距离。
序列选取原则
分子进化的研究对象是核酸和蛋白质序列。如果要研究某个基因的进化,是应该选用它的 DNA 序列,还是翻译后的蛋白质序列呢?
序列的选取要遵循以下原则:
1)如果 DNA 序列两两间的一致度≥70%,选用 DNA 序列。因为,如果 DNA 序列都如此相似,它们对应的蛋白质序列会相似到几乎看不出区别。这对于构建系统发生树是不利的。所以这种情况选用 DNA 序列,而不选蛋白质序列。
2)如果 DNA 序列两两间的一致度<70%,DNA 序列和蛋白质序列都可以选用。
4.5 MEGA构建NJ树
建树准备
这一节将给你演示如何用 MEGA7 软件构建系统发生树。为何挑这个软件来演示呢?
1) 软件免费;2)软件在默认设置下建树的效果就很好;3)软件被业界普遍认可,做出结果可 以用于文章发表;4)软件支持多操作系统,而且安装简单。
MEGA7 是完全的图形化界面操作(http://www.megasoftware.net/)。在接下来的例子里我们要为附件中 TIR.fasta 里的序列构建 NJ 树。TIR.fasta 里存储了 10 条人的不同 Toll 样受 胞内域的氨基酸序列。只有具有一定亲缘关系,也就是彼此比较相似,但又存在一定差别的序列拿来做多序列比对,或拿来构建系统发生树才有意义。
- 点击 MEGA 主窗口上的 File - Open A File - 找到并打开 TIR.fasta。这时,MEGA 会询问以何种方式打开。在介绍多序列比对时曾经说过,想要做系统发生树先要做多序列比对,然后把多序列比对结果提交给建树软件去建树。所以,此时可以输入一个现有的多序列比对,也可以输入原始序列,让 MEGA 先来做多序列比对,再建树。TIR.fasta 里面的序列为原始序列,需要 MEGA 来做多序列比对,因此选“Align”。
- 选择“Align”之后,在弹出的 Alignment Explorer 窗口上点击 Alignment - Align by ClustalW。MEGA 提供 ClustalW 和 Muscle 两种多序列比对方法。这里选择熟悉的 ClustalW 方法。弹出窗 口 询 问 “ Nothing selected for alignment. Select all? (是否要选择所有序列来做多序列比对) ”,选择 OK。
- 之后,弹出多序列比对参数设置窗口。这个窗口和EMBL 的多序列比对在线工具一样,可以设置替换记分矩阵、不同的空位罚分等参数。MEGA 的所有默认参数都不是随便设置的,这些经过反复考量默认设置好的参数保证了 MEGA 傻瓜机全自动档的品质。所以,当你无从下手的时候,直接点 OK,接受这些默认参数,开 始计算多序列比对。
将这个多序列比对作为中间结果保存下来。在 Alignment Explorer 窗口上点 Data - Export Alignment - MEGA Format。注意这里一定选 MEGA format 以方便 MEGA 继续加工。其他格式适用于其他软件。选择 MEGA 格式文件,文件自动赋予后缀名“.meg”。保存后,弹出窗口,要求给这组数据命名,命名为“test”。生成的 TIR.meg 文件属纯文本文件,可以用记事本打开查看,里面是用 MEGA 固有格式书写的多序列比对结果。
可以双击“TIR.meg”文件将其导入 MEGA。也可直接将其拖入 MEGA 主窗口中。拖入后, 主窗口里增加了一个TA图标。点击该图标,弹出新窗口 Sequence Data Explorer,其中显示 出 MEGA 格式的多序列比对。
再点击 Sequence Data Explorer 窗口上的 TA 按钮。点击后, 多序列比对中的 10 条序列之上增加了一行。这一行是根据多序列比对结果分析得出共有序列(consensus sequence),也就是每一列里出现次数最多的那个字母。多序列比对中,每一 列里的字母如果和共有序列的字母相同则打点,不同则标出不同的字母,空位还是空位。
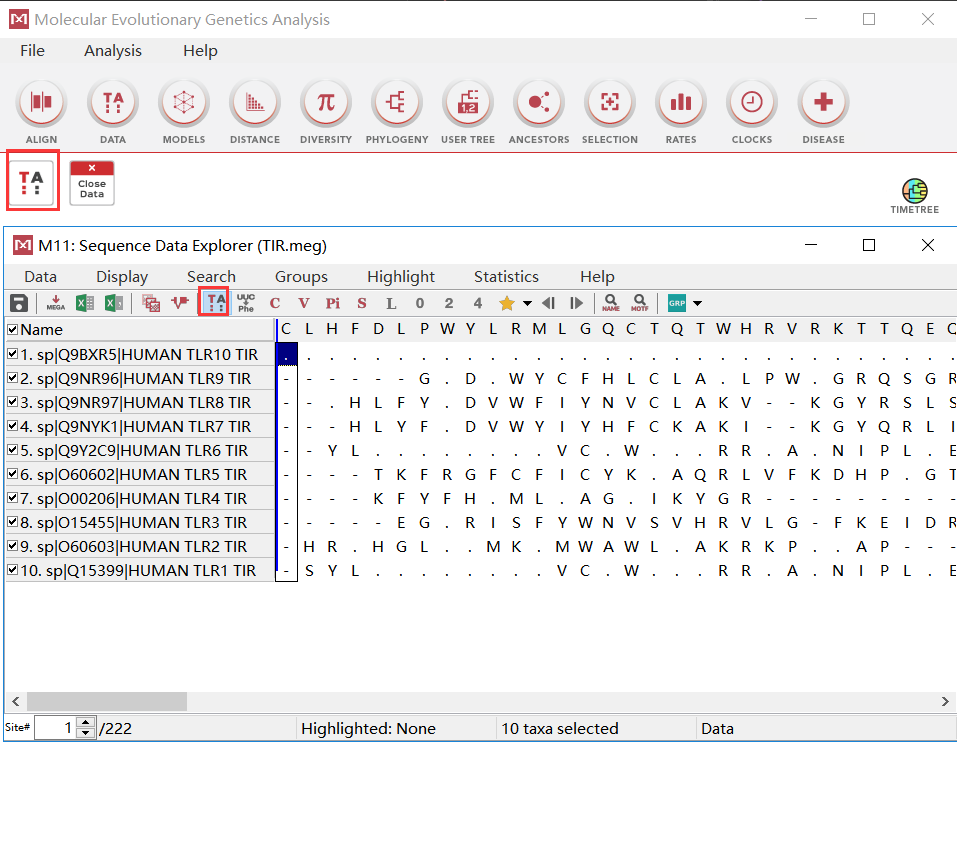
如果你想进一步了解这些序列的保守程度,可以点击C按钮,以黄色标记保守列,或者点击V按钮,以标黄标记不保守列。通过对比对结果的进一步分析,可以淘汰掉一些序列。比如海选的序列里有有一 些明显不合群的序列,就可以把它们前面的钩去掉,不让它们参与建树,以免影响建树质量。
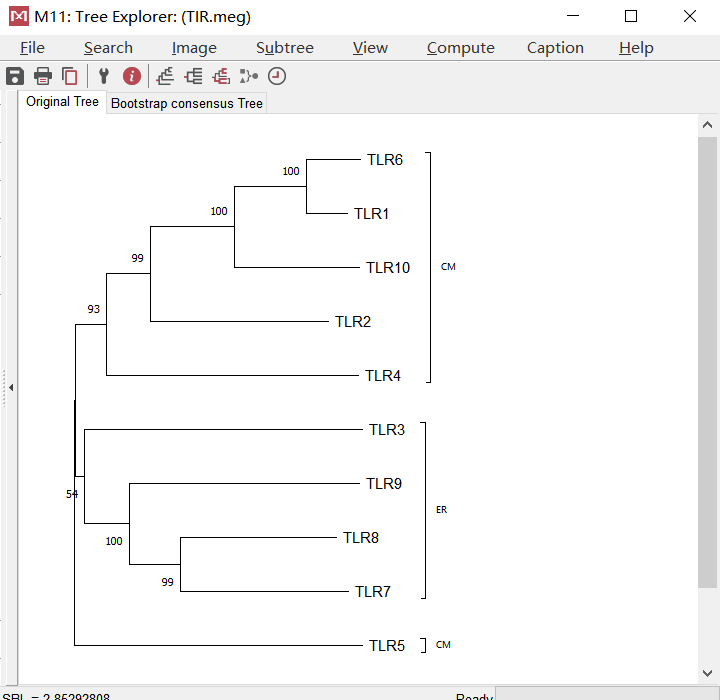
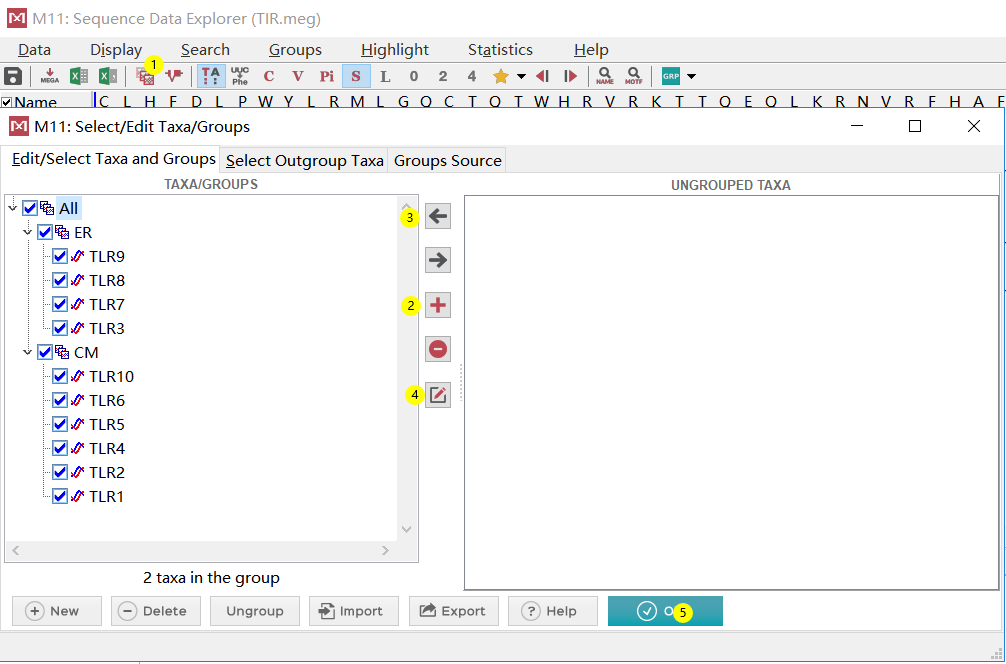
此外,还可以对这些序列进行分组标记。点击分组按钮“①”。我们让 TLR3、7、8、9这四个内质网上的蛋白质序列在一组,点 “②,更改组名为“ER”。然后按住 Ctrl 键同时选中 Ungrouped Taxa 列表里的 TLR3、7、8、9 四条序列的名字。选中后点击”③“ ,将选中的序列归入 ER 组。再次点击“②” ,创建第二组,更名为 CM,即细胞膜上的蛋白质。按住 Ctrl 选中 Ungrouped Taxa 列表里剩下的所有序列,点击”③” ,将它们归入 CM 组。当序列数量较多的时候,人为分组,可以从树上更加清楚的看出组内哪些成员叛逃了去了别组。
此外,输入序列的名字较长。这样的名字如果作为构建的系统发生树上叶子的名字,会破坏树的外观也不利于信息的解读。因为,需要人为修改一下序列的名字。选中序列后点击“④”,把名字改为能区分彼此的关键词,比如只保留 TLR*。全部更改好之后,点 “⑤”,准备工作全部完成。
建树过程
开始建树。点击 MEGA 主窗口上的 Phylogeny 下拉菜单,之前提到的各种建树方法这里基本都有。选 Neighbor Joining(最近邻居法)。弹出窗口询问是否使用当前 TIR.meg 里面的数据,选 Yes。接下来,弹出参数设置窗口(Analysis Preferences)。
参数设置对构建的系统 发生树的准确程度来说非常重要。在树构建好之后,还经常需要根据树的具体情况,重新设置参数,并重新建树,如此反复,直至结果令人满意为止。同样的,如果对于参数依然摸 着头脑,不知道应该怎样设置,那么继续使用 MEGA 的自动挡,接受默认参数,也能做出基本满意的系统发生树。如果想要做出更好的树,至少应该掌握其中三个参数的设置。
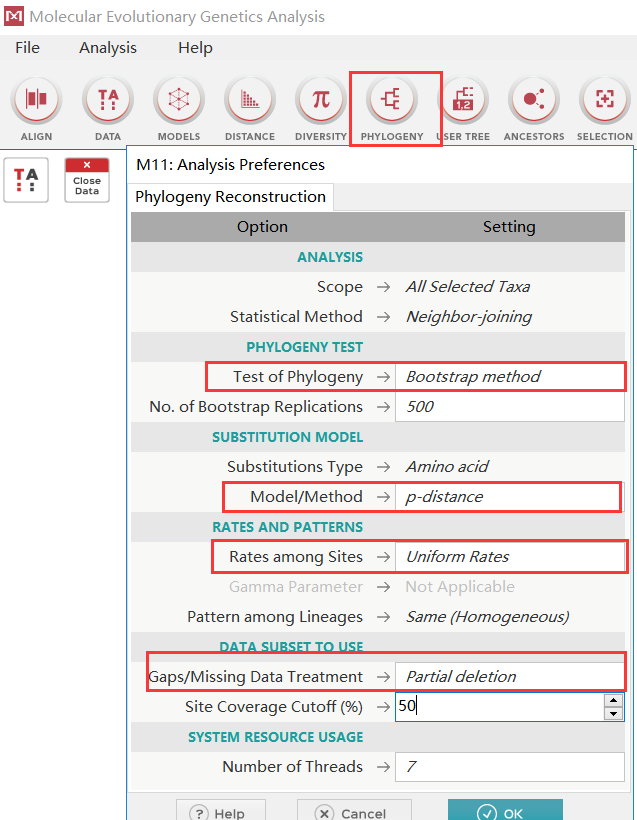
第一个参数 是 Test of phylogeny(建树的检验方法)。默认值为不进行检验。这显然比较偷工减料,不检验怎么知 道建出来的树质量如何,是否可信!因此,检 验 方 法 选 常 用 的Bootstrp method(步长检验)。步长检验需要设定检验次数,通常为100 的倍数,比如设置为 500。
步长检验是根据所选的建树方法,计算并绘制指定次数株系统发生树。因为大多数建树方法的核心算法都是统计概率模型,所以每次计算出的树都会有所差别。而建好的系统发生树上每个节点上都会标记一个数字,它代表了指定次数次计算所得出的系统发生树中有百分之多少棵树都含有这一节点。一般来说,绝大多数节点上的数值都大于 70%的树才可信。个别低于 70%的节点可以暂且容忍,或通过添加,删减序列来改善质量。
第二个参数是,Substitution Model。它是选择计算遗传距离时使用的计算模型。理论上应该尝试各种模型,根据检验结果选择最合适的模型进行计算。但在实际操作中,可先尝试选用较简单的距离模型,比如 p-distance。
第三个参数是 Gap/Missing Data Treatment。大多数建树方法会要求删除多序列比对中含有空位的列。但是根据遗传距离度量方法的不同,删除原则也不同。如果是以序列间不同残 基的个数来度量遗传距离的话,这里需要选择 Complete deletion(全部删除)。如果是其他方 法,比如这里选用的 NJ 方法,可以选择 Partial deletion(部分删除)。删除程度定在 50%,即,保留一半含有空位的列。
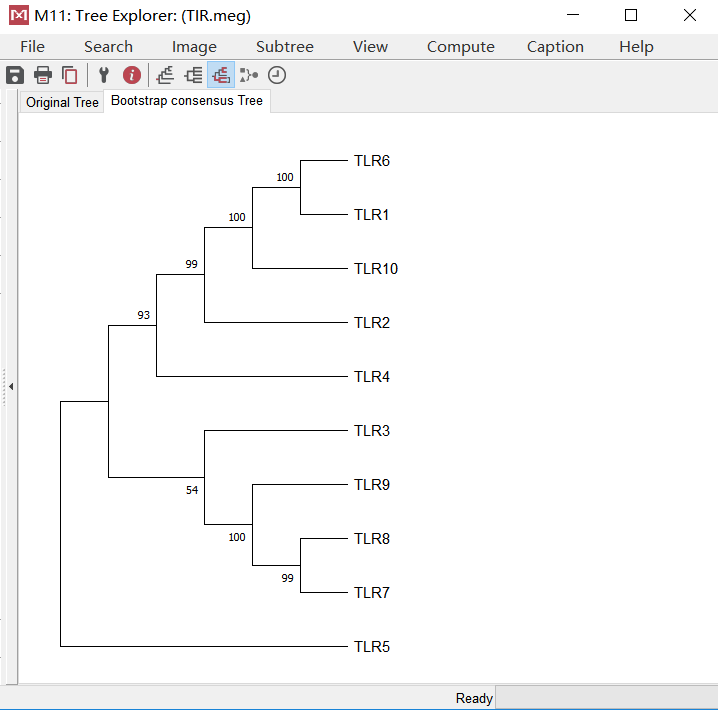
按照以上方案参数设置后,点击“OK”开始构建系统发生树。经过一番计算之后,新窗口 Tree Explorer 里展示的就是创建好的系统发生树。这个窗口里有两个标签页。第一个是 Original Tree(原始树),第二个是 Bootstrap consensus tree(步长检验合并出来的树)。Bootstrap consensus tree 上,节点处的数字表示,经步长检验有百分之几的树具有这根树枝,即,反应了该树枝的可信度。当前构建的这株系统发生树中,绝大多数节点处的数值都是≥70 的,所以这株树整体上是可信的。
Original Tree 是步长检验构建的 500 株树中的一株,未经过多棵树合并,所以树枝的长短可以精确代表遗传距离。此外, 从这株树也可以看出之前的人为分组情况是不是发生了意想不到的变化。比如,TLR5 似乎脱离了CM 组,成为了外类群,从而确定了树根。